Перейти к:
Оценка тропизма и биораспределения синтетических и природных аденоассоциированных вирусных векторов in vivo методом секвенирования нового поколения
https://doi.org/10.30895/2221-996X-2024-24-2-215-228
Резюме
ВВЕДЕНИЕ. Создание синтетических векторов на основе аденоассоциированных вирусов (ААВ) при разработке препаратов для генной терапии является трудоемким и дорогостоящим процессом. Для ускорения и снижения стоимости разработки генотерапевтических препаратов оптимальным представляется совершенствование методов оценки биораспределения и эффективности трансдукции in vivo ААВ векторов.
ЦЕЛЬ. Разработка методики биоинформатической оценки библиотек синтетических векторов на основе ААВ для анализа биораспределения и эффективности трансдукции ААВ in vivo.
МАТЕРИАЛЫ И МЕТОДЫ. Конструировали синтетические ААВ векторы путем добавления специальной метки (баркода), специфичной для каждого исследуемого серотипа ААВ — 12-нуклеотидной последовательности, фланкированной с 5'-конца кодирующей последовательностью репортерного белка (зеленый флуоресцентный белок). Плазмиды с уникальными баркодами нарабатывали в компетентных клетках Escherichia coli XL10-Gold и использовали для сборки ААВ-библиотек: ААВ-библиотека L1 — с количеством вирусных геномов (вг) 10¹⁰; библиотека L2 с количеством вг — 10¹¹. Для продукции ААВ проводили трансфекцию клеток НЕК293Т. Векторы в виде двух ААВ-библиотек вводили внутривенно мышам линии С57В1/6N. Из органов и тканей животных выделяли ДНК и РНК для последующего анализа методом секвенирования нового поколения. Анализировали количество ДНК- и РНК-баркодов в органах мышей для оценки биораспределения и эффективности трансдукции ААВ. Баркоды идентифицировали с помощью выравнивания на ожидаемые последовательности, затем подсчитывали их количество и нормировали на количество в исходной библиотеке.
РЕЗУЛЬТАТЫ. Созданы 7 вирусных конструкций на основе ААВ различных серотипов, 6 из которых — синтетические (sААВ1, sААВ2, sААВ3, sААВ4, sААВ5, sААВ6), в составе двух ААВ-библиотек. При проведении секвенирования образцов из органов мышей обнаружено значимое количество ДНК-баркодов обеих ААВ-библиотек в образцах всех органов, за исключением мозга. Для библиотеки L1 детектированы РНК-баркоды на достаточном уровне в четырех органах: скелетная мускулатура, сердце, печень и надпочечники; для библиотеки L2 — дополнительно к перечисленным органам в гонадах и почках. При анализе эффективности трансдукции (по уровню РНК-баркодов с поправкой на ДНК-баркоды) показано, что наиболее перспективными вариантами для генной терапии заболеваний печени является sAAВ5, а для заболеваний надпочечников — sAAВ2 и sAAВ6.
ВЫВОДЫ. Разработанная методика биоинформатической оценки библиотек синтетических векторов на основе ААВ позволяет проводить анализ биораспределения и эффективности трансдукции ААВ векторов в организме. Предложенный подход является перспективным для подбора оптимальных ААВ векторов для конкретных органов и тканей при разработке генотерапевтических препаратов.
Ключевые слова
Для цитирования:
Максимов Д.О., Наумова Д.А., Астахова Е.А., Артемьев В.В., Бирюков С.А., Абрамов И.С., Навойкова А.А., Рудев Н.В., Феоктистова С.Г., Глазова О.В., Митяева О.Н., Волчков П.Ю. Оценка тропизма и биораспределения синтетических и природных аденоассоциированных вирусных векторов in vivo методом секвенирования нового поколения. БИОпрепараты. Профилактика, диагностика, лечение. 2024;24(2):215-228. https://doi.org/10.30895/2221-996X-2024-24-2-215-228
For citation:
Maksimov D.O., Naumova D.A., Astakhova E.A., Artemev V.V., Biryukov S.A., Abramov I.S., Navoikova A.A., Rudev N.V., Feoktistova S.G., Glazova O.V., Mityaeva O.N., Volchkov P.Yu. In vivo evaluation of tropism and biodistribution of synthetic and natural adeno-associated viral vectors by next-generation sequencing. Biological Products. Prevention, Diagnosis, Treatment. 2024;24(2):215-228. (In Russ.) https://doi.org/10.30895/2221-996X-2024-24-2-215-228
ВВЕДЕНИЕ
Векторы на основе аденоассоциированных вирусов (ААВ) являются перспективным инструментом для генной терапии благодаря способности эффективно доставлять генетический материал в клетки-мишени и низкой иммуногенности [1]. Разные серотипы ААВ имеют различные показатели эффективности трансдукции и биораспределения в органах и тканях [2–4]. Наиболее перспективны для генной терапии те серотипы ААВ, которые селективно трансдуцируют целевые органы-мишени, не затрагивая остальные ткани [5]. Разработка синтетических векторов на основе серотипов ААВ актуальна для достижения оптимальных показателей биораспределения и трансдукции, а также для снижения риска нежелательных токсических эффектов, в частности иммунных реакций [6–9].
Внесение изменений в гены, кодирующие белки капсида ААВ, при создании синтетических векторов влияет на сродство капсида к рецепторам клеток и, следовательно, на параметры биораспределения вируса в организме [10–12]. В связи с этим при создании синтетических ААВ необходимо проводить их тестирование для изучения показателей биораспределения в тканях и эффективности трансдукции. Для исследования этих показателей наиболее информативными являются методы с использованием животных моделей: биоимиджинг в живом организме; определение сигнала от репортера в извлеченных органах и тканях; количественное определение вирусных частиц (например, с помощью количественной ПЦР) [13]. Реализация этих подходов подразумевает необходимость тестирования на животных каждого синтетического вектора на основе ААВ по отдельности, что является ресурсо- и трудозатратным процессом и препятствует ускоренной разработке векторов для генной терапии [14][15].
Для решения описанной проблемы авторами данной работы предлагается создание высокопроизводительной скрининговой системы на основе ААВ-библиотек, состоящих из нескольких серотипов ААВ, что позволит быстро отбирать варианты ААВ с интересующими характеристиками биораспределения и трансдукции.
В настоящее время существует несколько систем молекулярного баркодирования ААВ-библиотек [16]. В работе K. Adachi с соавт. [17] впервые показано применение технологии баркодирования ДНК с последующим использованием метода секвенирования нового поколения (next-generation sequencing, NGS) в отношении ААВ. Полученные результаты позволили построить функциональную карту капсида ААВ, которая выявила аминокислотные замены, влияющие на функциональную активность капсида. Подход, направленный на анализ биораспределения ААВ в организме с использованием технологии баркодирования ААВ-библиотек, был впервые предложен сотрудниками лаборатории С. Золотухина (Университет Флориды, США) [18]. Анализ биораспределения ААВ в тканях после инъекции проводится по оценке ДНК, а анализ тропизма — способности экспрессировать трансген в тканях, регистрируется по оценке уровня РНК [19].
Несмотря на значительный прогресс в области разработки методов для массовой характеризации вариантов капсидов ААВ с помощью NGS-секвенирования [16–18], биоинформатические подходы для анализа данных молекулярного баркодирования в тканях организма остаются несовершенными. В данной работе применялась обобщенная линейная регрессия, что позволило учесть тот факт, что для различных повторов неизбежно получается разное покрытие, а значит и разная достоверность. Это учитывается моделью при оценке среднего. Кроме того, важным новым аспектом данного исследования являлась оценка эффективности трансдукции (тропизм с учетом биораспределения), так как эта оценка имеет важное значение для подбора стратегии проведения генотерапии. Так, например, если вариант вируса обладает высокой эффективностью трансдукции, но из-за особенностей профиля биораспределения в малой степени попадает в целевую ткань, то в этом случае локальная инъекция препарата вируса может быть более предпочтительной.
Цель работы — разработка методики биоинформатической оценки библиотек синтетических векторов на основе ААВ для анализа биораспределения и эффективности трансдукции ААВ in vivo.
МАТЕРИАЛЫ И МЕТОДЫ
Дизайн эксперимента
Схема дизайна эксперимента представлена на рисунке 1.
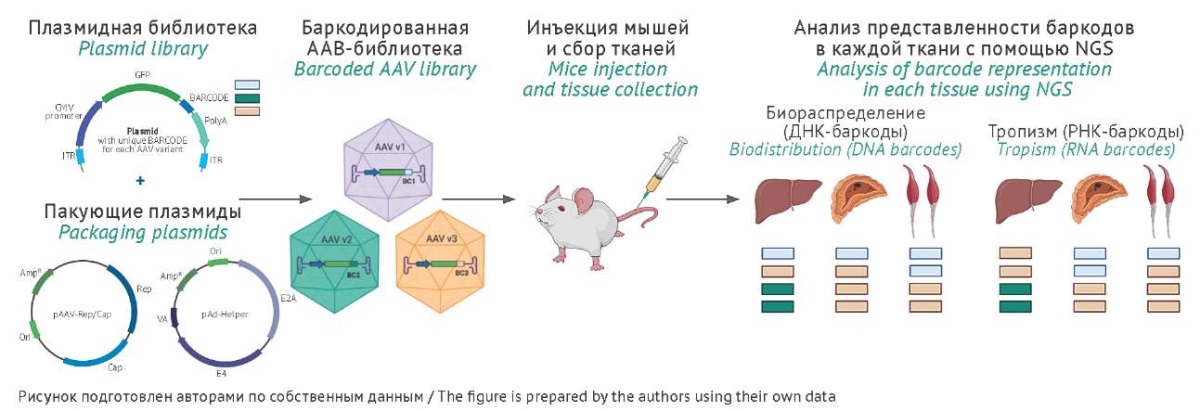
Рис. 1. Дизайн эксперимента. ААВ — аденоассоциированный вирус, NGS — секвенирование нового поколения.
Fig. 1. Experiment design. AAV, adeno-associated virus; NGS, next-generation sequencing.
Баркодирование ААВ-библиотек
Для изучения свойств векторов на основе различных серотипов ААВ проводили добавление специальной метки (баркода), специфичной для каждого исследуемого серотипа ААВ (табл. 1). Баркод представлял собой 12-нуклеотидную последовательность, фланкированную с 5'-конца кодирующей последовательностью репортерного белка (улучшенный зеленый флуоресцентный белок, enhanced green fluorescent protein, EGFP) и с 3'-конца сигналом полиаденилирования мРНК (poly(A) signal) (рис. 2).
Таблица 1. Серотипы аденоассоциированных вирусных векторов (ААВ)
и использованные для них баркоды
Table 1. Serotypes of adeno-associated virus (AAV) vectors and the corresponding barcodes
ААВ-библиотека AAV library | Серотип ААВ AAV serotype | Последовательность ДНК баркода (5'–3') DNA barcode sequence (5'–3') |
L1, L2 | sААВ1/sAAV1 | TTT GTA GTC GAC |
L1, L2 | sААВ2/sAAV2 | TCA CCT CAA GAC |
L1, L2 | sААВ3/sAAV3 | GTG AGA TAA TGT |
L1, L2 | sААВ4/sAAV4 | ATC ATA AAA GTT |
L1 | sААВ5/sAAV5 | CAT GAT AAT ATG |
L1, L2 | sААВ6/sAAV6 | TCG ACC TCC CCG |
L1 | ААВ2/AAV2 | GCA ATT GTG ATG |
Таблица составлена авторами / The table is prepared by the authors

Рис. 2. Общая схема баркодирования плазмид. ITR — инвертированные терминальные повторы; CMV — промотор цитомегаловируса; EGFP — улучшенный зеленый флуоресцентный белок; barcode — баркод; poly(A) — сигнал полиаденилирования; Pr F — прямой праймер; Pr R + 12n barcode — обратный праймер с последовательностью баркода.
General plasmid barcoding scheme. ITR, inverted terminal repeats; CMV, cytomegalovirus promoter; poly(A), polyadenylation signal; Pr F, forward primer; Pr R + 12n barcode, reverse primer and barcode sequence.
Для баркодирования плазмид предварительно амплифицировали репортерный ген (eGFP) с использованием Q5® High-Fidelity 2X Master Mix (NEB, США) согласно рекомендациям производителя с помощью пула праймеров, из которых обратные праймеры дополнительно несли различные последовательности баркода. Амплифицированные фрагменты клонировали по методу Гибсона с использованием NEBuilder HiFi DNA Assembly Master Mix (NEB, США) согласно рекомендациям производителя. Полученными плазмидами, содержащими репортерный ген с баркодом, трансформировали компетентные клетки Escherichia coli XL10-Gold (Aligent) и рассевали их на чашки Петри с агаризованной селективной средой LB (Диаэм, Россия) с антибиотиком. Далее проводили скрининг колоний для подтверждения правильности целевой вставки, после чего наращивали клеточную массу в жидкой питательной среде LB (Диаэм, Россия), выделяли плазмидную ДНК и проводили рестрикционный анализ. Плазмиды, соответствующие требуемой рестрикционной карте, секвенировали (при участии ЗАО «Евроген», Россия). Всего было собрано 30 конструкций. Плазмиды с уникальными баркодами использовались для сборки ААВ-библиотек: ААВ-библиотека L1 — с количеством вирусных геномов (вг) 10¹⁰; библиотека L2 с количеством вг — 10¹¹.
Сборка ААВ векторов
Для продукции ААВ проводили трансфекцию клеток НЕК293Т в присутствии PEI 25K (Polysciences, Индия) тремя плазмидами, несущими следующие гены: 1 — целевой ген eGFP; 2 — гены rep и cap ААВ (Cell Biolabs, США); 3 — гены-помощники ААВ (гены аденовируса E4, E2, VA) (Cell Biolabs, США) в соотношении 1:1:1 [20].
Подготовка и проверка плазмид выполнялась по стандартным протоколам культивирования и трансформации E. coli. После трансфекции и инкубации клетки лизировали в щелочном буфере в присутствии бензоназы (ГенТерра, Россия). Очистку клеточного лизата проводили в градиенте йодоксанола (60, 40, 25, 15%) (Sigma-Aldrich, CША) с последующим ультрацентрифугированием на центрифуге Optima XPN-80 Ultracentrifuge (Beckman Coulter, США) в течение 90 мин при температуре 10 °С и 360000 g, с последующей стерилизацией фильтрованием.
Для подсчета количества выделенного ААВ (вг/мкл) использовали количественную полимеразную цепную реакцию (qPCR) со специфическими к инвертированным терминальным повторам праймерами (fwd ITR primer, 5'-GGAACCCCTAGTGATGGAGTT; rev ITR primer, 5'-CGGCCTCAGTGAGCGA) с красителем Sybr Green (ЗАО «Евроген», Россия). В качестве стандартов использовали серию разведений лианеризованной плазмиды с трансгеном, упакованной в капсид ААВ. Перед началом реакции образец обрабатывали ДНКазой (NEB, США) и подвергали последующему термическому воздействию для разрушения капсидов (95 °С, 10 мин).
Исследование с экспериментальными животными
Исследование проводили на мышах-самцах линии C57Bl/6N в возрасте 7 нед. (животные предоставлены НПП «Питомник лабораторных животных» Филиала ФГБУН Института биоорганической химии им. академиков М.М. Шемякина и Ю.А. Овчинникова РАН). Протокол исследования с использованием лабораторных животных был одобрен локальным Биоэтическим комитетом Московского государственного университета имени М.В. Ломоносова (№ 62-д-з от 16.05.2023). Все эксперименты проводили в соответствии с требованиями Директивы 2010/63/EU Европейского парламента и Совета Европейского союза от 22.09.2010 по охране животных, используемых в научных целях1.
Было сформировано 3 группы животных, в каждой из которых было по 4 мыши: контрольная группа и две экспериментальные. Животным из контрольной группы водили инъекционно PBS, а животным из экспериментальных групп проводили инъекции ААВ-библиотек (L1 и L2).
Подготовка препарата ААВ и инъекции производились в асептических условиях. Препарат разводили в PBS, конечный объем для инъекции составлял 50 мкл. Готовили растворы ААВ для введения мышам в дозах: векторы ААВ-библиотеки L1 — концентрация 10¹⁰ вг/животное, ААВ-библиотеки L2 — концентрация 10¹¹ вг/животное. Инъекция проводилась в хвостовую вену со скоростью 1 мл/мин.
Через месяц после инъекций животных подвергали эвтаназии методом дислокации шейных позвонков. Проводили отбор следующих органов: гонады, легкие, головной мозг, скелетная мускулатура, надпочечники, печень, почки, селезенка, сердце, сетчатка глаза, тимус. После извлечения органы помещались в пробирки типа «Эппендорф» объемом 2 мл для последующего анализа.
Подготовка библиотек ДНК и кДНК
ДНК и РНК из органов мышей выделяли с помощью набора для параллельного выделения нуклеиновых кислот HiPure DNA/RNA kit (Magen, Китай). Концентрацию нуклеиновых кислот и наличие примесей оценивали с использованием спектрофотометра NanoDrop™ 2000c (Thermo, США). В качестве опытных образцов для анализа использовали образцы нуклеиновых кислот, выделенных из органов: 88 образцов РНК, 88 образцов ДНК. В качестве контрольных образцов использовали: образцы ААВ-библиотек L1 и L2, которые не вводили животным; отрицательный контроль — 4 образца ДНК и 4 образца РНК, выделенные из органов животных, которым вводили PBS без ААВ; отрицательные контроли всех стадий пробоподготовки — обратная транскрипция (ОТ), 1 стадия ПЦР, 2 стадия ПЦР.
Для получения кДНК использовали набор HiScript III 1st Strand cDNA Synthesis Kit (+gDNA wiper) для синтеза кДНК и удаления геномной ДНК (Vazyme, Китай). Для реакции использовали 1 мкг РНК каждого образца; проводили постановку 1 раунда ПЦР. Праймеры были сконструированы таким образом, чтобы 3'-концы были комплементарны фланкирующей баркод области GFP, а 5'-концы несли последовательности универсальных адаптеров, аналогичных последовательностям набора Nextera XT DNA Library Prep Kit (Illumina, США).
ПЦР проводили с помощью Q5® High-Fidelity PCR Kit (NEB, США) согласно инструкции производителя. Количество циклов амплификации составляло 20 и было определено в рамках контрольных экспериментов с помощью постановки ПЦР в реальном времени — 200 нг геномной ДНК, выделенной из разных органов, в присутствии флуоресцентного красителя EvaGreen (Biotium, США). Для образцов всех органов показатель количества циклов амплификации (Cq) не превышал 20.
Секвенирование
Секвенирование полученных библиотек проводили с использованием платформы MiSeq (Illumina, США) с помощью набора реагентов MiSeq Reagent Kit v2 (300 cycles).
Биоинформатический анализ результатов секвенирования
Парные чтения, полученные после секвенирования в формате fastq, были картированы на референс с помощью программы minimap2 v.2.26-r1175 [21]. Референс содержал последовательность исходной плазмиды, кодирующей последовательность EGFP и регуляторных элементов (EGFP, ITR), в котором последовательность баркода длиной 12 нуклеотидов кодировалась с помощью 12 символов N (табл. 1). Полученные bam-файлы были отсортированы и индексированы с помощью программы Samtools v.1.172, а затем анализировались с использованием программного обеспечения Jupyter Notebook (python v.3.10.11, библиотеки pysam v.0.21.0, biopython v.1.81 [22]). Сначала выделялись участки чтений, картированные на баркод, затем проводилось попарное выравнивание каждого из этих участков (с длиной больше 5 нуклеотидов) с каждой из референсных последовательностей баркодов с помощью класса PairwiseAligner (параметры scoring = «blastn», mode = «global») из модуля Align пакета Biopython3. Чтобы отсечь возможные баркоды низкого качества, было сгенерировано 100 случайных баркодов и осуществлялось выравнивание с каждым из этих вариантов. В случае если участок чтения имел скор выравнивания (числовая оценка, отражающая степень сходства между двумя последовательностями) на баркод вируса выше, чем на случайный, то вариант вируса считался детектированным. Для каждой библиотеки (ДНК исходной ААВ-библиотеки; ДНК или РНК, выделенные из ткани или органа мыши) оценивалось количество каждого из вариантов вируса, при этом в случае, когда левое и правое прочтение детектировали отличающиеся баркоды (таких оказалось всего 3 пары чтений для всех библиотек), пара исключалась из рассмотрения.
Оценка относительного количества баркодов в ААВ-библиотеке
Оценка относительного количества производилась независимо для каждого органа или ткани с учетом распределения вирусов (баркодов) исходной ААВ-библиотеки. Анализ проводился в RStudio (R v.4.3.1)4 c помощью пакетов stats v.4.3.1 и ggplot2 v.3.4.2. Оценка числа детектированных баркодов проводилась с помощью обобщенной линейной модели (функция glm, family = «poisson») по формуле (1):
B ≈ V + L + ln(Nткань) + ln(NLвирус/NLобщ) – 1, (1)
где, B — число детектированных баркодов конкретного варианта вируса (число ДНК-баркодов при оценке биораспределения; число РНК-баркодов при оценке тропизма); V — категориальная переменная, которая отражает эффективность варианта вируса в ткани или органе (усредненную по двум ААВ-библиотекам); L — поправка на ААВ-библиотеку (L1 — 10¹⁰ вг, L2 — 10¹¹ вг); Nткань — общее количество баркодов, детектированных в образце ткани; NLвирус и NLобщ — количество баркодов варианта вируса и общее количество баркодов, соответственно, в исходной ААВ-библиотеке.
Эффективность трансдукции — способность вируса проникать в клетки и приводить к экспрессии трансгена — величина, представляющая собой тропизм с коррекцией на биораспределение, а в логарифмическом масштабе — разность между вирусной нагрузкой на ткань и показателем эффективности трансдукции вирусом этой ткани. Для лучшей оценки эффективности трансдукции, позволяющей определить стандартную ошибку как в отдельных дата поинтах, так и среднюю, применялась следующая формула (2):
B ≈ V + RNAV + L + ln(Nткань) + ln(NLвирус/NLобщ) – 1, (2)
где B — число детектированных баркодов конкретного варианта вируса (ДНК-баркоды и РНК-баркоды варианта вируса в ткани); V и RNAV — категориальные переменные, отражающие биораспределение и эффективность трансдукции, соответственно, серотипа вируса в ткани/органе (усредненную по двум библиотекам). Переменная RNAV имеет ненулевое значение, только когда B — РНК-баркоды (для ДНК-баркодов всех вирусов переменная имеет одно значение — «DNA»). Параметры L, Nткань, NLвирус и NLобщ те же, что и в предыдущей модели.
При сравнении вирусов между собой проводилась оценка отдельно для каждой библиотеки и мыши (без параметра L в формуле, с оценкой параметра V на соответствующем подмножестве данных). Стандартные ошибки оценки параметров определяли с помощью функции «summary» в RStudio (R v.4.3.1)5. При графической визуализации значения биораспределения (рис. 5, 6) или тропизма (рис. 7, 8) менее минус 5 были обрезаны до этой точки, а стандартная ошибка установлена как 0. В каждом графике (диаграмма «ящик с усами») (рис. 3–10) использовали следующие элементы: центральная линия — медиана; границы «ящика» — верхний и нижний квартили; черные линии («усы») — 1,5-интерквартильный размах; вертикальные красные линии — стандартная ошибка среднего; черные точки — статистические выбросы.
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
Сборка баркодированных ААВ-библиотек
Секвенирование показало, что 15 из 30 отправленных на секвенирование плазмид, кодирующих EGFP, содержали подтвержденные 12-нуклеотидные баркоды. Среди этих 15 образцов плазмид не было обнаружено повторения баркода. Далее из этих подтвержденных секвенированием образцов были отобраны 7 плазмид с баркодами, которые в дальнейшем использовались для сборки различных серотипов ААВ векторов (табл. 1).
Указанные серотипы использовались для составления двух ААВ-библиотек (L1 и L2): L1 включала в себя 7 серотипов ААВ, L2 — 5 серотипов (табл. 1). Для составления обеих библиотек использовались различные серотипы векторов в равном соотношении. Для инъекционного введения мышам использовали количество вирусных геномов, равное в случае ААВ-библиотеки L1 — 10¹⁰ вг на животное, для L2 — 10¹¹ вг на животное.
Контроль качества ААВ-библиотек для секвенирования
Перед пробоподготовкой проводилась спектрофотометрическая оценка качества ДНК и РНК по показателю соотношений длин волн. Значения поглощения (A) 260/280 и 260/230 нм соответствовали приемлемым для ампликоновой пробоподготовки образцов высокопроизводительного секвенирования (1,8–2,0 и 2,0–2,2 соответственно). Финальный контроль размера ААВ-библиотек проводили с помощью капиллярного электрофреза (биоаналайзера) Qsep 100 (BiOptic, Китай). Размер ААВ-библиотек соответствовал ожидаемым для продукта второго раунда амплификации (продукт 1 раунда + размер индекс-адаптерных последовательностей) с учетом погрешности измерения. В результате оптимизированной пробоподготовки удалось избавиться от коротких димеров праймеров и адаптеров (~150 по), критически влияющих на расчет реакции секвенирования и дальнейший анализ данных.
Анализ биораспределения и тропизма
В связи с тем что активность используемого в данной работе CMV промотора (рис. 2) варьирует в различных тканях организма, подход, предложенный авторами, заключался в сравнении биораспределения и тропизма различных серотипов ААВ в пределах отдельной ткани.
В настоящей работе термин «биораспределение» используется для описания способности вируса достигать ткани и оценивается по количеству ДНК. Достижение вирусом ткани не означает его проникновение в клетки, так как вирусы могут находиться на поверхности клеток. Термин «тропизм» в данной работе используется для описания способности вируса заражать ткань (достигать клетки, проникать в ядро и приводить к экспрессии трансгена) и оценивается по количеству РНК. Термин «эффективность трансдукции» обозначает способность вируса, достигнув ткани, заразить ее, и оценивается по количеству РНК с поправкой на количество ДНК вируса в ткани.
В рамках данного подхода количественное сравнение биораспределения и тропизма используемых серотипов ААВ между различными тканями/органами представлялось невозможным.
Для исследования биораспределения оценивалось количество вирусных ДНК-баркодов в целевых тканях и органах мышей (как описано в разделе «Материалы и методы»). По результатам анализа секвенирования удалось обнаружить ДНК-баркоды каждой из ААВ-библиотек в каждом из проанализированных образцов органов/тканей (рис. 3). Полученный результат свидетельствует о том, что количество вирусной ДНК в каждом образце оказалось достаточным для проведения амплификации и дальнейшего секвенирования ампликонов.
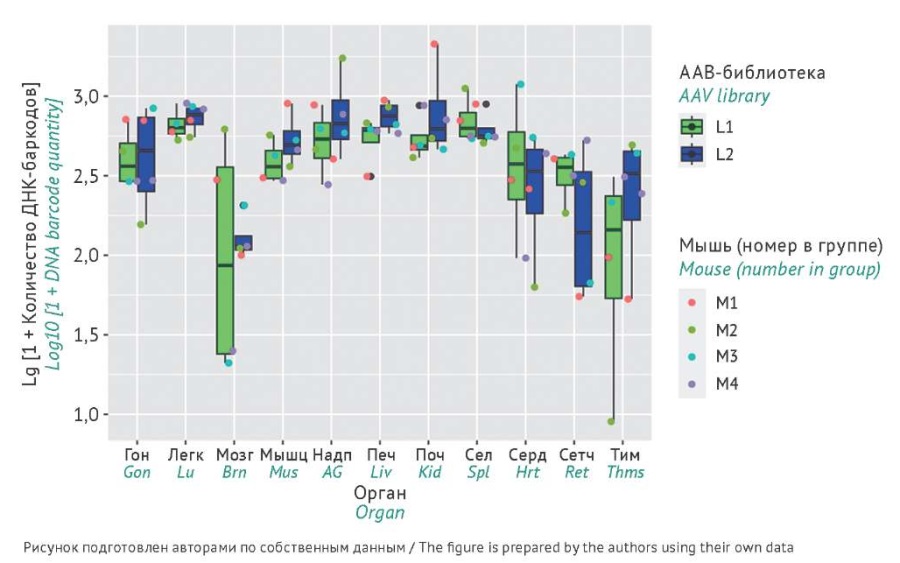
Рис. 3. Биораспределение аденоассоциированного вируса (ААВ) в образцах органов и тканей мышей по количеству ДНК-баркодов после инъекционного введения ААВ-библиотек L1 или L2. По оси Х указаны органы/ткани: Гон — гонады, Легк — легкие, Мозг — головной мозг, Мышц — скелетная мускулатура, Надп — надпочечники, Печ — печень, Поч — почка, Сел — селезенка, Серд — сердце, Сетч — сетчатка глаза, Тим — тимус. По оси Y указан десятичный логарифм количества ДНК-баркодов каждой из ААВ-библиотек в каждом из проанализированных образцов органов/тканей. В легенде указаны ААВ-библиотеки и порядковый номер мыши в группе (для каждой экспериментальной группы n=4). Черными линиями показан 1,5-интерквартильный размах.
Fig. 3. Adeno-associated virus (AAV) biodistribution in mouse organs and tissues by the quantity of DNA barcodes after injection of AAV libraries L1 or L2. The X-axis indicates organs/tissues: Gon, gonads; Lu, lungs; Brn, brain; Mus, skeletal muscles; AG, adrenal glands; Liv, liver; Kid, kidney; Spl, spleen; Hrt, heart; Ret, retina; Thms, thymus. The Y-axis indicates the decimal logarithm of the quantity of DNA barcodes from each AAV library in each of the organ/tissue samples analysed. The legend indicates the AAV library and the sequential number of the mouse in the group (for each experimental group, n=4). Black lines show the 1.5-interquartile range.
Следует отметить, что количество ДНК-баркодов в образцах мозга мышей было достаточное для анализа, однако оно было меньше (статистически значимо) относительно среднего числа ДНК-баркодов, обнаруженных в других органах (p=0,002, двусторонний тест Вилкоксона). Вероятно, обнаруженные в образцах мозга ДНК-баркоды являются остаточной ДНК, локализованной на стенках сосудов головного мозга мышей. Таким образом, протестированные в данной работе серотипы ААВ не способны преодолевать гематоэнцефалический барьер (ГЭБ).
Для исследования тропизма проводилась оценка количества вирусных РНК-баркодов в целевых тканях и органах мышей (рис. 4). Был установлен порог минимального количества детектированных баркодов, равный 10 (красная пунктирная линия на рис. 4), и в рассмотрение включались только те ткани, для которых нижний квартиль числа детектированных баркодов был выше 1. Таким образом, для ААВ-библиотеки L1, содержащей меньшее количество ААВ (10¹⁰ вг), удалось детектировать РНК-баркоды на достаточном уровне только в случае четырех тканей: скелетная мускулатура, сердце, печень и надпочечники. Для ААВ-библиотеки L2, содержащей 10¹¹ вг, как и ожидалось, эффективная трансдукция по детектируемому количеству РНК-баркодов (у всех мышей группы) зарегистрирована в большем количестве органов/тканей — дополнительно к вышеуказанным, в почках и гонадах (рис. 4).
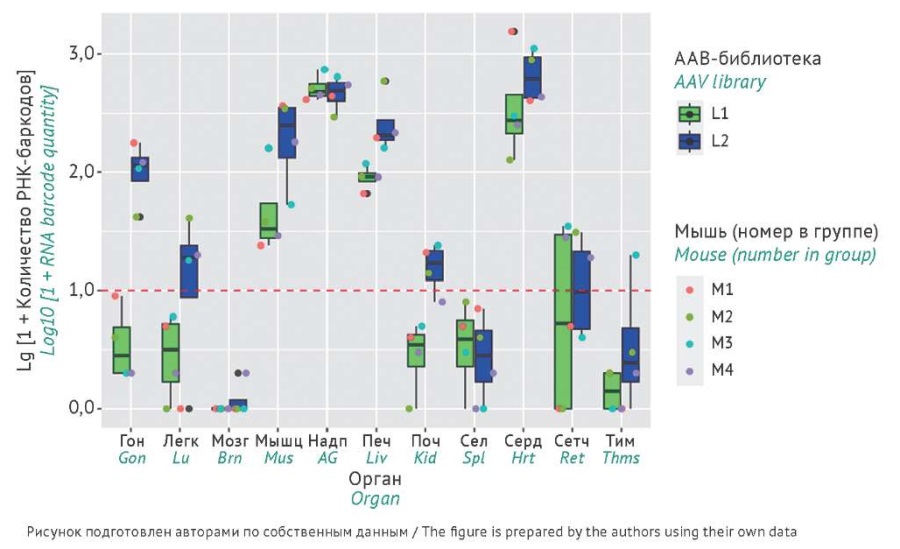
Рис. 4. Тропизм вариантов аденоассоциированного вируса (ААВ) к органам и тканям мышей после инъекционного введения ААВ-библиотек L1 или L2. По оси Х указаны органы/ткани: Гон — гонады, Легк — легкие, Мозг — головной мозг, Мышц — скелетная мускулатура, Надп — надпочечники, Печ — печень, Поч — почка, Сел — селезенка, Серд — сердце, Сетч — сетчатка глаза, Тим — тимус. По оси Y указан десятичный логарифм количества РНК-баркодов каждой из ААВ-библиотек в каждом из проанализированных образцов органов/тканей. В легенде указаны ААВ-библиотеки и порядковый номер мыши в группе (для каждой экспериментальной группы n=4). Черными линиями показан 1,5-интерквартильный размах. Красная пунктирная линия соответствует 9 детектированным баркодам — условная отсечка минимального количества баркодов, допускающая сравнение между серотипами вируса.
Fig. 4. Tropism of adeno-associated virus (AAV) variants to mouse organs and tissues after injection of AAV libraries L1 or L2. The X-axis indicates organs/tissues: Gon, gonads; Lu, lungs; Brn, brain; Mus, skeletal muscles; AG, adrenal glands; Liv, liver; Kid, kidney; Spl, spleen; Hrt, heart; Ret, retina; Thms, thymus. The Y-axis indicates the decimal logarithm of the quantity of RNA barcodes from each AAV library in each of the organ/tissue samples analysed. The legend indicates the AAV library and the sequential number of the mouse in the group (for each experimental group, n=4). Black lines show the 1.5-interquartile range. The red dotted line corresponds to 9 detected barcodes and represents a cutoff for the minimum quantity of barcodes that allows for comparison between virus serotypes.
Показано практически полное отсутствие экспрессии вирусной РНК в образцах головного мозга и селезенки мышей при использовании обеих ААВ-библиотек. В случае головного мозга отсутствие экспрессии связано, по-видимому, с неспособностью используемых ААВ преодолевать ГЭБ; в случае селезенки возможно влияние других факторов, так как проникновение ААВ в селезенку было подтверждено высоким содержанием ДНК-баркодов в органе.
Анализ биораспределения и тропизма в печени и надпочечниках
Результаты анализа биораспределения показывают высокую согласованность результатов как внутри, так и между ААВ-библиотеками (рис. 5, 6), что позволяет определить серотипы ААВ, способные наиболее эффективно достигать печени и надпочечников (sААВ5 и sААВ2 соответственно).
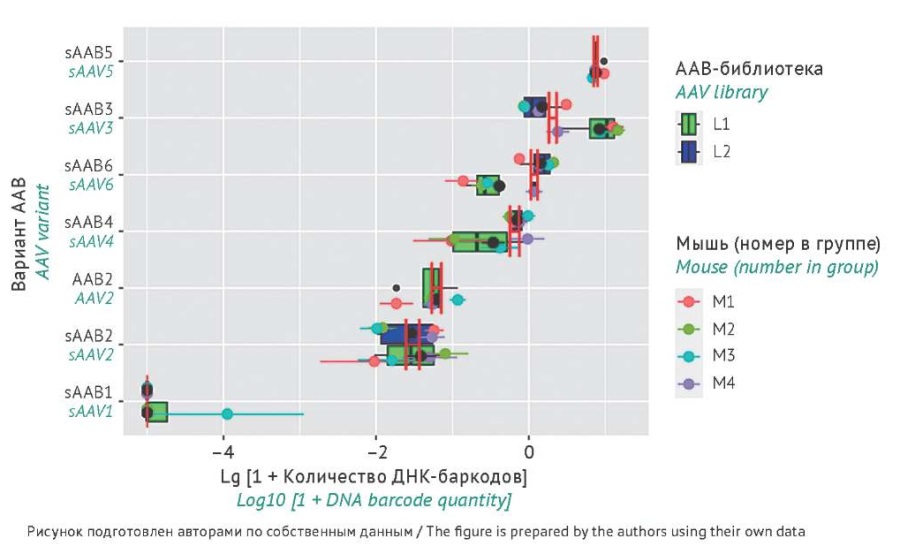
Рис. 5. Количество детектированных ДНК-баркодов в образцах печени после инъекционного введения мышам библиотек аденоассоциированного вируса (ААВ) L1 или L2. Вертикальные красные линии отражают стандартную ошибку среднего для каждого из серотипов ААВ. В легенде указаны ААВ-библиотеки и порядковый номер мыши в группе (для каждой экспериментальной группы n=4). Серотипы вирусов по оси Y упорядочены по убыванию вирусной нагрузки.
Fig. 5. Quantity of detected DNA barcodes in liver samples after injection of adeno-associated virus (AAV) libraries L1 or L2 into mice. Vertical red lines represent the standard error of the mean for each AAV serotype. The legend indicates the AAV library and the sequential number of the mouse in the group (for each experimental group, n=4). The Y-axis lists virus serotypes in descending order by viral load.
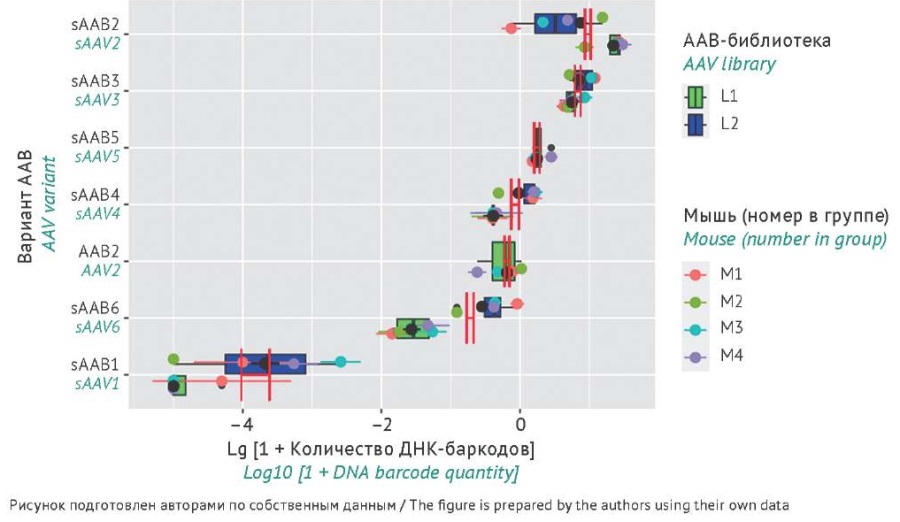
Рис. 6. Количество детектированных ДНК-баркодов в образцах надпочечников после инъекционного введения мышам библиотек аденоассоциированного вируса (ААВ) L1 или L2. Вертикальные красные линии отражают стандартную ошибку среднего для каждого из серотипов ААВ. В легенде указаны ААВ-библиотеки и порядковый номер мыши в группе (для каждой экспериментальной группы n=4). Серотипы вирусов по оси Y упорядочены по убыванию вирусной нагрузки.
Fig. 6. Quantity of detected DNA barcodes in adrenal samples after injection of adeno-associated virus (AAV) libraries L1 or L2 into mice. Vertical red lines represent the standard error of the mean for each AAV serotype. The legend indicates the AAV library and the sequential number of the mouse in the group (for each experimental group, n=4). The Y-axis lists virus serotypes in descending order by viral load.
Результаты анализа тропизма продемонстрировали высокую согласованность для большей части серотипов ААВ (рис. 7, 8). Следует отметить, что для серотипа sAAВ2 в надпочечниках и печени имеется значительное расхождение результатов между библиотеками L1 и L2, что, возможно, объясняется небольшим количеством повторов.
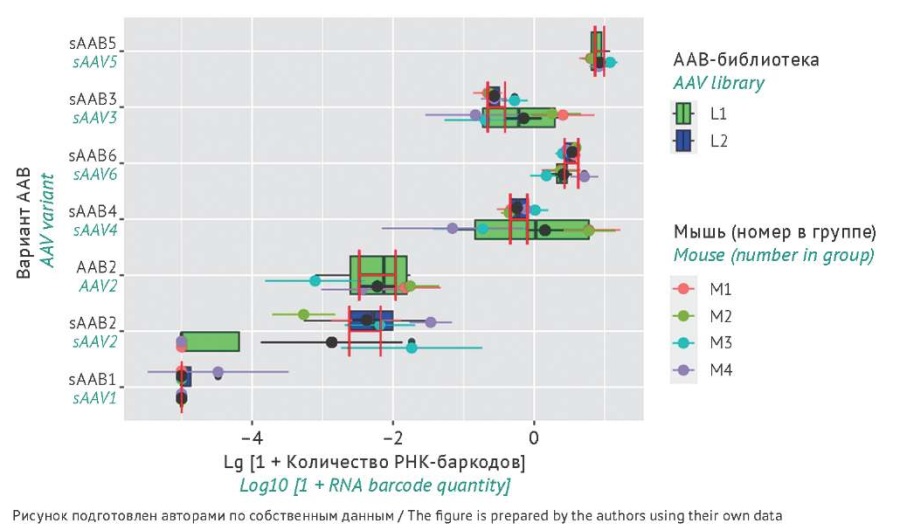
Рис. 7. Количество детектированных РНК-баркодов в образцах печени после инъекционного введения мышам библиотек аденоассоциированного вируса (ААВ) L1 или L2. Вертикальные красные полосы отражают стандартную ошибку среднего для каждого из серотипов ААВ. В легенде указаны ААВ-библиотеки и порядковый номер мыши в группе (для каждой экспериментальной группы n=4). Серотипы вирусов по оси Y упорядочены как на рис. 5.
Fig. 7. Quantity of detected RNA barcodes in liver samples after injection of adeno-associated virus (AAV) libraries L1 or L2 into mice. Vertical red lines represent the standard error of the mean for each AAV serotype. The legend indicates the AAV library and the sequential number of the mouse in the group (for each experimental group, n=4). Virus serotypes on the Y-axis are ordered as in Fig. 5.
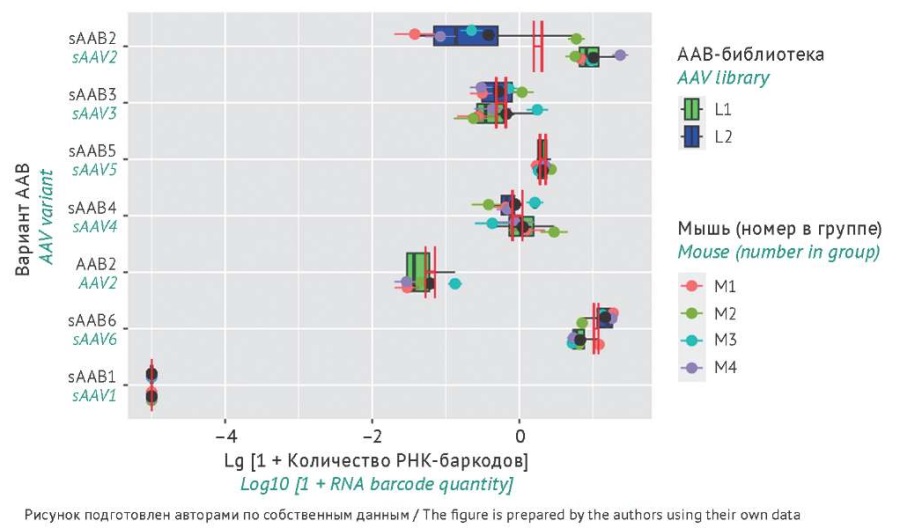
Рис. 8. Количество детектированных РНК-баркодов в образцах надпочечников после инъекционного введения мышам библиотек аденоассоциированного вируса (ААВ) L1 или L2. Вертикальные красные полосы отражают стандартную ошибку среднего для каждого из серотипов ААВ. В легенде указаны ААВ-библиотеки и порядковый номер мыши в группе (для каждой экспериментальной группы n=4). Серотипы вирусов по оси Y упорядочены как на рис. 6.
Fig. 8. Quantity of detected RNA barcodes in adrenal samples after injection of adeno-associated virus (AAV) libraries L1 or L2 into mice. Vertical red lines represent the standard error of the mean for each AAV serotype. The legend indicates the AAV library and the sequential number of the mouse in the group (for each experimental group, n=4). Virus serotypes on the Y-axis are ordered as in Fig. 6.
Оценка тропизма имеет большое прикладное значение для выбора серотипа ААВ, позволяющего добиться наибольшей экспрессии трансгена в ткани-мишени. Так, среди протестированных серотипов ААВ наиболее перспективным для генной терапии заболеваний, связанных с печенью (например, гемофилии A, B и C), является sAAВ5 (рис. 7), а для заболеваний, связанных с надпочечниками (например, для врожденной дисфункции коры надпочечников) — sAAВ2 и sAAВ6 (рис. 8).
Анализ эффективности трансдукции в печени и надпочечниках
Численные значения, полученные в результате оценки биораспределения и тропизма, представлены в логарифмическом масштабе, поэтому положительные значения указывают на повышенную эффективность трансдукции относительно ожидаемой, а отрицательные — на пониженную.
На рисунке 9 (опубликован на сайте журнала6) можно видеть, что серотип sAAВ3 обладает пониженной эффективностью трансдукции в печени относительно ожидаемой. Не удалось выявить варианты ААВ, обладающие достоверно повышенной эффективностью трансдукцией печени. Так, несмотря на положительные медианные значения эффективности трансдукции у sААВ1, наблюдались большие разбросы между образцами, полученными от разных животных. Вариант sAAВ6 обладал повышенной относительно ожидаемой в 100 раз эффективностью трансдукции в надпочечниках (рис. 10, опубликован на сайте журнала7), что делает его одним из наиболее эффективных для этого органа, несмотря на низкое количество ДНК (рис. 6). Представленные результаты указывают на принципиальную важность проведения исследований по характеризации ААВ-библиотек не только на уровне ДНК, но и на уровне матричной РНК.
Результаты оценки биораспределения и эффективности трансдукции in vivo при использовании ААВ векторов
Разработанная методика оценки биораспределения, тропизма и эффективности трансдукции ААВ-библиотек позволяет эффективно выявлять серотипы ААВ, наиболее перспективные для терапии конкретного органа или ткани. Однако получаемые численные значения, характеризующие эти параметры, могут быть сравнены друг с другом только в рамках конкретного органа или ткани, но не между ними. Это является ограничением данной работы. В связи с этим в дальнейших исследованиях планируется оценивать абсолютные количества вирусной ДНК и РНК с помощью количественной ПЦР с нормализацией на единицу генома клетки и экспрессию генов домашнего хозяйства (например, GAPDH) соответственно. Однако различные ткани обладают разными уровнями экспрессии GAPDH, что может привести к систематической ошибке. Несмотря на наличие различных подходов к характеризации ААВ, описанных в литературе [16–19], эта проблема остается нерешенной.
На рисунках 11 и 12 (опубликованы на сайте журнала8) представлены обобщенные данные по биораспределению и тропизму вариантов ААВ в исследованных органах и тканях. При интерпретации графиков необходимо иметь в виду, что они построены на основе сравнения вариантов вирусов в каждом органе/ткани независимо. Графики не отражают абсолютных значений, поэтому прямое сравнение значений по отдельным органам между собой невозможно. Из представленных данных видно, что некоторые серотипы ААВ обладают широким биораспределением и тропизмом (например, sААВ5, рис. 119), в то время как другие обладают относительной специфичностью, например sААВ2 более эффективен для трансдукции в надпочечниках (рис. 1110). В целом серотипы с широким биораспределением демонстрируют и широкий тропизм.
ВЫВОДЫ
- Разработан метод биоинформатической оценки библиотек синтетических векторов на основе аденоассоциированного вируса различных серотипов для исследования биораспределения и эффективности трансдукции in vivo. Предложенный метод характеризуется высокой производительностью, экономичностью и возможностью быстрого отбора векторов с желаемыми характеристиками для проведения генной терапии, что позволит ускорить и оптимизировать разработку генотерапевтических препаратов.
- Показана согласованность результатов как внутри, так и между библиотеками синтетических векторов, что имеет большое значение с точки зрения верификации методики. Однако требуется проведение дальнейших исследований на большем массиве данных.
- Выявлены серотипы аденоассоциированного вируса с наиболее оптимальными характеристиками биораспределения и эффективности трансдукции in vivo. Так, при анализе эффективности трансдукции показано, что наиболее перспективным для генной терапии заболеваний печени является sAAВ5, а для заболеваний надпочечников — sAAВ2 и sAAВ6.
Вклад авторов. Все авторы подтверждают соответствие своего авторства критериям ICMJE. Наибольший вклад распределен следующим образом: Д.О. Максимов — концепция работы, обработка экспериментальных результатов, подготовка иллюстративного материала; Д.А. Наумова — анализ и систематизация источников литературы, формулировка выводов; Е.А. Астахова — анализ источников литературы, участие в формулировке выводов; В.В. Артемьев — анализ источников литературы, сборка баркодированных конструкций; С.А. Бирюков — сборка векторов на основе аденоассоциированного вируса; И.С. Абрамов — проведение секвенирования; A.A. Навойкова — подготовка плазмид, отработка методов; Н.В. Рудев — проведение исследований с лабораторными животными; С.Г. Феоктистова — сборка баркодированных конструкций, подготовка рукописи; О.В. Глазова — организация и проведение экспериментов с животными, забор биоматериала, выделение и анализ нуклеиновых кислот; О.Н. Митяева — планирование экспериментов; утверждение окончательной версии рукописи для публикации; П.Ю. Волчков — утверждение окончательной версии рукописи для публикации.
Соответствие принципам этики. Экспериментальные исследования с животными проводились в соответствии с Директивой 2010/63/EU. Протокол исследования был одобрен локальным Биоэтическим комитетом Московского государственного университета имени М.В. Ломоносова (№ 62-д-з от 16.05.2023).
Authors’ contributions. All the authors confirm that they meet the ICMJE criteria for authorship. The most significant contributions were as follows. D.O. Maksimov conceptualised the study, processed the experimental results, and prepared the illustrative material. D.A. Naumova analysed and collated literature and formulated the conclusions. E.A. Astakhova analysed literature and formulated the conclusions. V.V. Artemev analysed literature and assembled the barcoded constructs. S.A. Biryukov assembled the AAV vectors. I.S. Abramov performed sequencing. A.A. Navoikova prepared the plasmids and refined the methods. N.V. Rudev conducted the experiments with laboratory animals. S.G. Feoktistova assembled the barcoded constructs and prepared the manuscript. O.V. Glazova organised and conducted the experiments with laboratory animals, collected biomaterial specimens, isolated and analysed nucleic acids. O.N. Mityaeva planned the experiments, approved the final version of the manuscript for publication. P.Yu. Volchkov approved the final version of the manuscript for publication.
Ethics approval. The animals were housed and used for experimental procedures in compliance with Directive 2010/63/EU and the recommendations of the Local Bioethics Committee, Lomonosov Moscow State University (Ethics Approval 62-d-z of 16.05.2023).
1. Directive 2010/63/EU of the European Parliament and of the Council of 22 September 2010 on the protection of animals used for scientific purposes. https://eur-lex.europa.eu/eli/dir/2010/63/oj
2. https://www.htslib.org/
3. https://biopython.org/
4. https://posit.co/download/rstudio-desktop/
5. Там же.
6. https://doi.org/10.30895/2221-996X-2024-24-2-215-228-suppl
7. Там же.
8. https://doi.org/10.30895/2221-996X-2024-24-2-215-228-suppl
9. https://doi.org/10.30895/2221-996X-2024-24-2-215-228-suppl
10. Там же.
Список литературы
1. Wang D, Tai PWL, Gao G. Adeno-associated virus vector as a platform for gene therapy delivery. Nat Rev Drug Discov. 2019;18(5):358–78. https://doi.org/10.1038/s41573-019-0012-9
2. Srivastava A. In vivo tissue-tropism of adeno-associated viral vectors. Curr Opin Virol. 2016;21:75–80. https://doi.org/10.1016/j.coviro.2016.08.003
3. Pañeda A, Vanrell L, Mauleon I, Crettaz JS, Berraondo P, Timmermans EJ, et al. Effect of adeno-associated virus serotype and genomic structure on liver transduction and biodistribution in mice of both genders. Hum Gene Ther. 2009;20(8):908–17. https://doi.org/10.1089/hum.2009.031
4. Hauck B, Xiao W. Characterization of tissue tropism determinants of adeno-associated virus type 1. J Virol. 2003;77(4):2768–74. https://doi.org/10.1128/jvi.77.4.2768-2774.2003
5. Han J, Zhu L, Zhang J, Guo L, Sun X, Huang C, et al. Rational engineering of adeno-associated virus capsid enhances human hepatocyte tropism and reduces immunogenicity. Cell Prolif. 2022;55(12):e13339. https://doi.org/10.1111/cpr.13339
6. Li C, Samulski RJ. Engineering adeno-associated virus vectors for gene therapy. Nat Rev Genet. 2020;21(4):255–72. https://doi.org/10.1038/s41576-019-0205-4
7. Rode L, Bär C, Groß S, Rossi A, Meumann N, Viereck J, et al. AAV capsid engineering identified two novel variants with improved in vivo tropism for cardiomyocytes. Mol Ther. 2022;30(12):3601–18. https://doi.org/10.1016/j.ymthe.2022.07.003
8. Ghauri MS, Ou L. AAV engineering for improving tropism to the central nervous system. Biology (Basel). 2023;12(2):186. https://doi.org/10.3390/biology12020186
9. Drouyer M, Chu TH, Labit E, Haase F, Navarro RG, Nazareth D, et al. Novel AAV variants with improved tropism for human Schwann cells. Mol Ther Methods Clin Dev. 2024;32(2):101234. https://doi.org/10.1016/j.omtm.2024.101234
10. Gigout L, Rebollo P, Clement N, Warrington KH, Muzyczka N, Linden RM, et al. Altering AAV tropism with mosaic viral capsids. Mol Ther. 2005;11(6):856–65. https://doi.org/10.1016/j.ymthe.2005.03.005
11. Wu Z, Asokan A, Grieger JC, Govindasamy L, AgbandjeMcKenna M, Samulski RJ. Single amino acid changes can influence titer, heparin binding, and tissue tropism in different adeno-associated virus serotypes. J Virol. 2006;80(22):11393–7. https://doi.org/10.1128/jvi.01288-06
12. Asokan A, Conway JC, Phillips JL, Li C, Hegge J, Sinnott R, et al. Reengineering a receptor footprint of adeno-associated virus enables selective and systemic gene transfer to muscle. Nat Biotechnol. 2010;28(1):79–82. https://doi.org/10.1038/nbt.1599
13. Gao GP, Alvira MR, Wang L, Calcedo R, Johnston J, Wilson JM. Novel adeno-associated viruses from rhesus monkeys as vectors for human gene therapy. Proc Natl Acad Sci USA. 2002;99(18):11854–9. https://doi.org/10.1073/pnas.182412299
14. Gao G, Vandenberghe LH, Alvira MR, Lu Y, Calcedo R, Zhou X, et al. Clades of adeno-associated viruses are widely disseminated in human tissues. J Virol. 2004;78(12):6381–8. https://doi.org/10.1128/jvi.78.12.6381-6388.2004
15. Zinn E, Pacouret S, Khaychuk V, Turunen HT, Carvalho LS, Andres-Mateos E, et al. In silico reconstruction of the viral evolutionary lineage yields a potent gene therapy vector. Cell Rep. 2015;12(6):1056–8. https://doi.org/10.1016/j.celrep.2015.07.019
16. Szumska J, Grimm D. Boosters for adeno-associated virus (AAV) vector (r)evolution. Cytotherapy. 2023;25(3):254–60. https://doi.org/10.1016/j.jcyt.2022.07.005
17. Adachi K, Enoki T, Kawano Y, Veraz M, Nakai H. Drawing a high-resolution functional map of adeno-associated virus capsid by massively parallel sequencing. Nat Commun. 2014;5:3075. https://doi.org/10.1038/ncomms4075
18. Marsic D, Govindasamy L, Currlin S, Markusic DM, Tseng YS, Herzog RW, et al. Vector design Tour de Force: integrating combinatorial and rational approaches to derive novel adeno-associated virus variants. Mol Ther. 2014;22(11):1900–9. https://doi.org/10.1038/mt.2014.139
19. Weinmann J, Weis S, Sippel J, Tulalamba W, Remes A, El Andari J, et al. Identification of a myotropic AAV by massively parallel in vivo evaluation of barcoded capsid variants. Nat Commun. 2020;11(1):5432. https://doi.org/10.1038/s41467-020-19230-w
20. Grimm D, Lee JS, Wang L, Desai T, Akache B, Storm TA, et al. In vitro and in vivo gene therapy vector evolution via multispecies interbreeding and retargeting of adeno-associated viruses. J Virol. 2008;82(12):5887–911. https://doi.org/10.1128/jvi.00254-08
21. Li H. Minimap2: Pairwise alignment for nucleotide sequences. Bioinformatics. 2018;34(18):3094–3100. https://doi.org/10.1093/bioinformatics/bty191
22. Cock PJA, Antao T, Chang JT, Chapman BA, Cox CJ, Dalke A, et al. Biopython: Freely available Python tools for computational molecular biology and bioinformatics. Bioinformatics. 2009;25(11):1422–3. https://doi.org/10.1093/bioinformatics/btp163
Об авторах
Д. О. МаксимовРоссия
Максимов Денис Олегович
Институтский переулок, д. 9, Долгопрудный, Московская область, 141700
Ленинские горы, д. 1, стр. 3, Москва, 119991
Балтийская, д. 8, Москва, 125315
Д. А. Наумова
Россия
Наумова Дарья Алексеевна
Институтский переулок, д. 9, Долгопрудный, Московская область, 141700
Е. А. Астахова
Россия
Астахова Екатерина Андреевна
Институтский переулок, д. 9, Долгопрудный, Московская область, 141700
В. В. Артемьев
Россия
Артемьев Валентин Владимирович
Институтский переулок, д. 9, Долгопрудный, Московская область, 141700
С. А. Бирюков
Россия
Бирюков Станислав Анатольевич, д-р физ.-мат. наук
Институтский переулок, д. 9, Долгопрудный, Московская область, 141700
И. С. Абрамов
Россия
Абрамов Иван Сергеевич
Институтский переулок, д. 9, Долгопрудный, Московская область, 141700
Ленинские горы, д. 1, стр. 3, Москва, 119991
ул. Балтийская, д. 8, Москва, 125315
А. А. Навойкова
Россия
Навойкова Анна Александровна
Институтский переулок, д. 9, Долгопрудный, Московская область, 141700
Н. В. Рудев
Россия
Рудев Николай Валерьевич
Институтский переулок, д. 9, Долгопрудный, Московская область, 141700
ул. Балтийская, д. 8, Москва, 125315
С. Г. Феоктистова
Россия
Феоктистова Софья Гумаровна
Институтский переулок, д. 9, Долгопрудный, Московская область, 141700
ул. Балтийская, д. 8, Москва, 125315
О. В. Глазова
Россия
Глазова Ольга Владимировна
Институтский переулок, д. 9, Долгопрудный, Московская область, 141700
ул. Балтийская, д. 8, Москва, 125315
О. Н. Митяева
Россия
Митяева Ольга Николаевна, канд. биол. наук
Институтский переулок, д. 9, Долгопрудный, Московская область, 141700
Ленинские горы, д. 1, стр. 3, Москва, 119991
ул. Балтийская, д. 8, Москва, 125315
П. Ю. Волчков
Россия
Волчков Павел Юрьевич, канд. биол. наук
Институтский переулок, д. 9, Долгопрудный, Московская область, 141700
Ленинские горы, д. 1, стр. 3, Москва, 119991
ул. Новогиреевская, д. 1, Москва, 111123
ул. Балтийская, д. 8, Москва, 125315
Дополнительные файлы

|
1. Рис. 9-12. | |
| Тема | ||
| Тип | Исследовательские инструменты | |
Скачать
(1MB)
|
Метаданные ▾ | |
Рецензия
Для цитирования:
Максимов Д.О., Наумова Д.А., Астахова Е.А., Артемьев В.В., Бирюков С.А., Абрамов И.С., Навойкова А.А., Рудев Н.В., Феоктистова С.Г., Глазова О.В., Митяева О.Н., Волчков П.Ю. Оценка тропизма и биораспределения синтетических и природных аденоассоциированных вирусных векторов in vivo методом секвенирования нового поколения. БИОпрепараты. Профилактика, диагностика, лечение. 2024;24(2):215-228. https://doi.org/10.30895/2221-996X-2024-24-2-215-228
For citation:
Maksimov D.O., Naumova D.A., Astakhova E.A., Artemev V.V., Biryukov S.A., Abramov I.S., Navoikova A.A., Rudev N.V., Feoktistova S.G., Glazova O.V., Mityaeva O.N., Volchkov P.Yu. In vivo evaluation of tropism and biodistribution of synthetic and natural adeno-associated viral vectors by next-generation sequencing. Biological Products. Prevention, Diagnosis, Treatment. 2024;24(2):215-228. (In Russ.) https://doi.org/10.30895/2221-996X-2024-24-2-215-228


































