Перейти к:
Проблема лекарственной устойчивости вируса SARS-CoV-2 к препаратам прямого противовирусного действия
https://doi.org/10.30895/2221-996X-2024-569
Резюме
ВВЕДЕНИЕ. Геном SARS-CoV-2 характеризуется высокой частотой мутаций, в связи с чем особое беспокойство вызывает проблема резистентности вируса к современным противови-
русным препаратам — ингибиторам основной протеазы (3CLpro) и РНК-зависимой РНК-полимеразы (RdRp).
ЦЕЛЬ. Проанализировать сведения о частоте встречаемости, спектре и причинах формирования мутаций лекарственной устойчивости вируса SARS-CoV-2 к ингибиторам основной протеазы и РНК-зависимой РНК-полимеразы, применяемым в клинической практике или находящимся в стадии разработки, на основе клинических, вирусологических и генотипических данных.
ОБСУЖДЕНИЕ. Анализ частоты встречаемости мутаций лекарственной устойчивости в геноме вируса SARS-CoV-2 к противовирусным препаратам (ингибиторы RdRp — ремдесивир и молнупиравир; ингибиторы протеазы 3CLpro — паксловид) за 2021–2024 гг. показал, что некоторые мутации существовали до начала использования препаратов. Уровень распространенности мутаций лекарственной резистентности в мире не превышает 0,5%. Однако согласно результатам клинических и экспериментальных исследований продемонстрирована возможность снижения восприимчивости к препаратам более чем в 200 раз, в том числе и с формированием мультирезистентных вариантов вируса SARS-CoV-2, что особенно актуально для пациентов с иммуносупрессией. У таких пациентов SARS-CoV-2 может сохраняться в течение многих месяцев, и противовирусный препарат может потерять свою эффективность по мере отбора или появления спонтанных мутаций, создавая риск распространения лекарственно-устойчивых штаммов и/или неблагоприятного исхода для пациентов.
ЗАКЛЮЧЕНИЕ. В связи с увеличением охвата лечением пациентов с COVID-19 существует риск роста резистентных вариантов вируса. Представленные данные свидетельствуют о необходимости проведения геномного эпидемиологического надзора с анализом потенциальных мишеней действия лекарственных препаратов с учетом данных клинического наблюдения пациентов, что в будущем может определять тактику терапии и необходимость разработки новых противовирусных препаратов (ингибиторов RdRp и протеаз), адаптированных к лекарственно-устойчивым вариантам SARS-CoV-2.
Ключевые слова
Для цитирования:
Гасич Е.Л., Кроткова Е.Н., Коско А.Д., Булда К.Ю., Красько А.Г., Дорофеева Е.А., Карпов И.А. Проблема лекарственной устойчивости вируса SARS-CoV-2 к препаратам прямого противовирусного действия. БИОпрепараты. Профилактика, диагностика, лечение. 2024;24(4):415-427. https://doi.org/10.30895/2221-996X-2024-569
For citation:
Gasich E.L., Krotkova E.N., Kasko A.D., Bulda K.Yu., Krasko A.G., Dorofeeva E.A., Karpov I.A. The problem of SARS-CoV-2 virus resistance to direct-acting antivirals. Biological Products. Prevention, Diagnosis, Treatment. 2024;24(4):415-427. (In Russ.) https://doi.org/10.30895/2221-996X-2024-569
ВВЕДЕНИЕ
Эпидемии и пандемии, вызванные возбудителями вирусных инфекций, являются одними из самых серьезных рисков для общественного здравоохранения. Ярким примером служит пандемия COVID-19, вызванная вирусом SARS-CoV-2, которая унесла жизни около 7 млн человек (данные на 01.08.2024)1. Первые случаи заболевания атипичной пневмонией были зарегистрированы в 2019 г. В качестве возбудителя был идентифицирован новый ранее неизвестный вирус, которому было присвоено название «коронавирус тяжелого острого респираторного синдрома 2» (SARS-CoV-2), а вызываемое им заболевание — «коронавирусная болезнь 2019» (COVID-19) [1]. Всемирная организация здравоохранения (ВОЗ) объявила 30 января 2020 г. вспышку коронавируса чрезвычайной ситуацией в области здравоохранения, имеющей международное значение2, а 11 марта 2020 г. — пандемией3.
Вирус SARS-CoV-2 претерпел значительные изменения за счет высокой скорости эволюции и преодоления межвидового барьера. Следовательно, с течением времени произошли изменения в генофонде популяции вируса. Об этом свидетельствует появление большого количества генетических вариантов, пять (альфа, бета, гамма, дельта, омикрон) из которых были классифицированы ВОЗ как варианты, вызывающие озабоченность4. В беспрецедентные сроки были разработаны и зарегистрированы вакцины против COVID-19 на основе как известных, так и новых биотехнологических платформ, и уже с 2021 г. началась всемирная кампания по массовой вакцинации [2]. Широкое применение вакцин сыграло важную роль в предупреждении смертности и госпитализации в результате заражения SARS-CoV-2 [3].
В связи с пандемией и высокой летальностью при COVID-19 начались и продолжаются до сих пор интенсивные исследования по поиску, разработке и применению эффективных противовирусных препаратов в отношении вируса SARS-CoV-2. До пандемии COVID-19 основное внимание исследователей и разработчиков уделялось вирусу иммунодефицита человека (ВИЧ) и вирусу гепатита С (ВГС), на долю которых приходилось более 67% одобренных противовирусных препаратов [4]. Обычно период от открытия возбудителя до выпуска специфичного в его отношении лекарственного препарата занимает длительное время. Впервые с применением трансляционных научных исследований удалось успешно провести кампанию по поиску лекарств среди применяющихся для лечения других возбудителей инфекционных заболеваний и разработке новых противовирусных препаратов в условиях быстро развивающейся пандемии [5].
При расширении масштабов эпидемии использование противовирусных препаратов для лечения коронавирусной инфекции может привести к новой проблеме, которая хорошо известна для таких вирусов, как вирус гепатита В, С, ВИЧ и вирус гриппа [6–9]. Это связано с естественной эволюцией вируса, в результате которой появляются нуклеотидные замены (мутации) в участках генома, кодирующих белки-мишени действия противовирусных препаратов. Как результат этих процессов — неэффективность терапии. В настоящее время для анализа резистентности, например, ВИЧ и ВГС, применяется в основном генотипирование, которое позволяет выявить мутации, ассоциированные с лекарственной устойчивостью как в случае вирусологического неуспеха к применяемой схеме лечения, так и в случае передачи устойчивого варианта вируса при любом способе заражения. Риск увеличения уровня резистентности в вирусной популяции повышается за счет более широкого применения противовирусных препаратов, монотерапии, неадекватного дозирования, длительного применения у пациентов с иммуносупрессией [10]. Критерий эффективности терапии — достижение вирусологического ответа, определяемого как отсутствие вирусной РНК в назофарингеальных мазках.
В настоящее время из большого количества препаратов для профилактики и лечения COVID-19, проверенных в доклинических и клинических исследованиях, три (ремдесивир, молнупиравир и паксловид) были одобрены или получили разрешение на экстренное использование в разных странах5. Как и другие противовирусные препараты, используемые для лечения РНК-вирусных инфекций, они нацелены на определенные этапы жизненного цикла вируса SARS-CoV-2 за счет подавления функций неструктурных белков. Выделяют две основные группы препаратов, действие которых направлено на разные мишени и механизмы: ингибиторы вирусной протеазы и РНК-зависимой РНК-полимеразы [11][12]. Поскольку геном SARS-CoV-2 характеризуется высокой частотой мутаций, особое беспокойство вызывает проблема резистентности вируса к имеющимся и разрабатываемым противовирусным препаратам.
Цель работы — проанализировать сведения о частоте встречаемости, спектре и причинах формирования мутаций лекарственной устойчивости вируса SARS-CoV-2 к ингибиторам основной протеазы и РНК-зависимой РНК-полимеразы, применяемым в клинической практике или находящимся в стадии разработки, на основе клинических, вирусологических и генотипических данных.
ОСНОВНАЯ ЧАСТЬ
Характеристика вируса SARS-CoV-2
Новый коронавирус SARS-CoV-2 относится к роду Betacoronavirus и содержит одноцепочечную плюс-цепь РНК размером около 30000 пар оснований, окруженную нуклеокапсидом и заключенную в фосфолипидную мембранную оболочку (рис. 1). Выход вируса из клетки осуществляется путем отпочковывания от мембраны клеток человека, поэтому по структуре оболочка вируса очень похожа на клеточную мембрану. Две трети вирусного генома кодируют два полипротеина, которые подвергаются протеолитическому процессингу с образованием 16 неструктурных белков (non-structural protein, nsp), тогда как оставшаяся область кодирует четыре структурных белка (спайковый, S; оболочечный, Е; мембранный, M; нуклеокапсидный, N) и девять вспомогательных белков. В оболочку встроены структурные гликопротеины в форме шипов (белок S), которые обеспечивают присоединение и слияние с мембраной клетки-хозяина [13]. Белок S связывается с ферментом ангиотензин-превращающего фермента типа 2 (АПФ2), который присутствует на поверхности мембраны эпителиальных клеток хозяина. Белок E взаимодействует с белками M и N в ходе сборки вируса. Всего на целый вирион приходится несколько копий этого белка. Пентамеры белка способны формировать ионные каналы и представляют собой важный фактор вирулентности SARS-CoV-2. Белок М играет ключевую роль в сборке вируса. Также этот белок способен изменять участок мембраны клетки и захватывать другие структурные белки, обеспечивая их взаимодействие в месте сборки вирусной частицы [14].
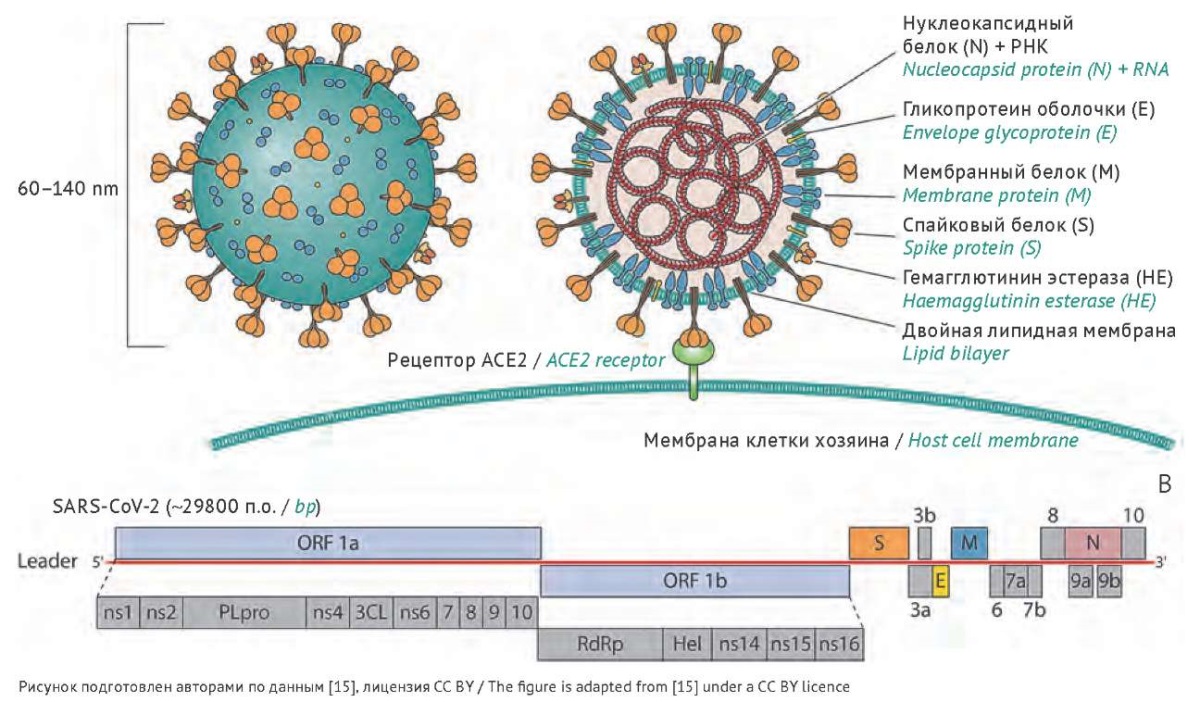
Рис. 1. Физическая и геномная структура SARS-CoV-2. А — схема вириона SARS-CoV-2; B — организация генома и белки с известными или неизвестными функциями.
Fig. 1. Physical and genome structure of SARS-CoV-2. A, SARS-CoV-2 virion diagram; B, genome organisation and proteins with known or unknown functions.
Неструктурные белки SARS-CoV-2 участвуют во многих биологических процессах, включая репликацию вирусного генома, транскрипцию и протеолиз. Некоторые из них выполняют функции ферментов: РНК-зависимая РНК-полимераза (RdRp), основная цистеиновая 3C-подобная протеаза (3CLpro) и папаин-подобная протеаза (PLpro) [15]. РНК-зависимая РНК-полимераза ответственна за синтез вирусной РНК. Протеаза 3CLpro осуществляет процессинг полипротеинов, транслируемых с вирусной РНК, распознавая специфические сайты расщепления. Неструктурный белок nsp3, содержащий домен PLpro, ответственен за расщепление вирусного полипротеина в ходе вирусного процессинга. Кроме того, nsp3/PLpro также расщепляет модификации убиквитина и убиквитинподобный белок внутри клетки-хозяина, нарушая тем самым развитие реакций врожденного иммунитета [16].
Основу для эволюции вируса SARS-CoV-2 составляют два события — появление мутаций и процесс рекомбинации. Известно, что частота и скорость появления мутаций для разных вирусов отличается. Так, для вируса гриппа H3N2 она составляет 1,35×10-5, для ВИЧ — 14×10-5 [17][18]. Скорость мутаций в геноме SARS-CoV-2 оценивается в 1×10-5 замен оснований в год [19]. Получены убедительные доказательства, что скорость эволюции вируса начала меняться с развитием пандемии COVID-19. Если в начале эпидемии средняя скорость эволюции генома SARS-CoV-2 варьировала в пределах от 1,78×10-3 до 1,83×10-3 замен оснований в год [20], то уже весной 2020 г. скорость эволюции SARS-CoV-2 составляла 4,1×10-4 замен оснований [21]. Большинство мутаций и рекомбинаций в ходе эволюции остаются нейтральными, но часть из них дает возбудителю определенные преимущества: более эффективное заражение чувствительных клеток, появление новых путей проникновения вируса в клетку, устойчивость к иммунобиологическим агентам и терапевтическим препаратам (лекарственная устойчивость).
Основные мишени действия противовирусных препаратов
С начала пандемии для терапии COVID-19 применяются противовирусные препараты, действие которых направлено на блокирование синтеза ключевых белков вируса и подавление его репликации [22]. Наиболее интенсивные исследования ведутся в направлении разработки ингибиторов RdRp и протеаз 3CLpro, которые играют ведущую роль в репродукции вируса SARS-CoV-2 и для которых определены кристаллические структуры, что позволяет осуществлять структурно-ориентированный дизайн лекарственных препаратов [23]. Мишени действия препаратов (РНК-зависимая РНК-полимераза и вирусные протеазы 3CLpro и PLpro) представлены на рисунке 2.
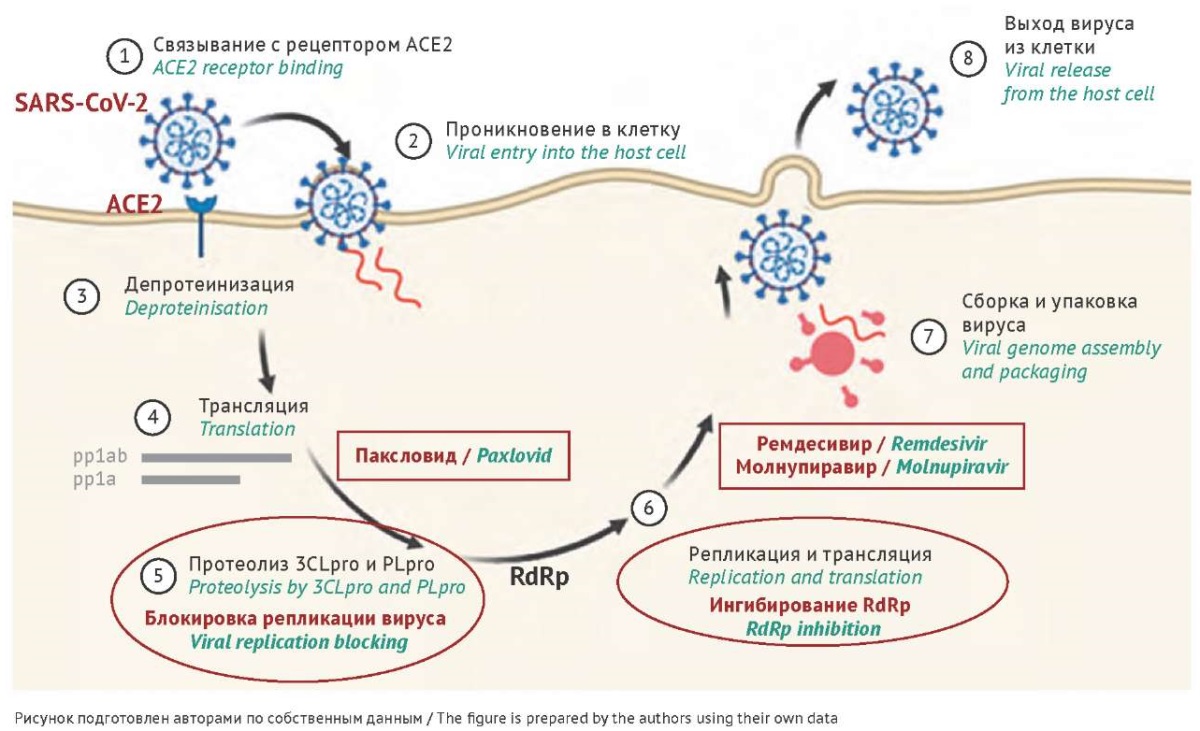
Рис. 2. Мишени действия препаратов на ключевых этапах жизненного цикла SARS-CoV-2, включая этапы: высвобождение вирусной РНК (депротеинизация, 3), трансляция вирусной РНК (4), протеолиз полипротеинов с участием 3CLpro и PLpro (5), репликация и трансляция (6), сборка и упаковка вирусных частиц (7), выход вириона из клетки (8).
Fig. 2. Targets for medicinal products at the key SARS-CoV-2 life cycle stages, including viral RNA release (deproteinisation (3)), viral RNA translation (4), proteolytic cleavage of polyproteins by 3CLpro and PLpro proteases (5), replication and translation (6), packaging and assembly of viral particles (7), and virion release from the host cell (8).
Ингибиторы РНК-зависимой РНК-полимеразы
Инициация репликации вируса и синтез субгеномных вирусных РНК с дальнейшим синтезом структурных и вспомогательных белков вируса начинается после трансляции кодируемой вирусом РНК-зависимой РНК-полимеразы (RdRp) с участием кофакторов nsp7 и nsp8 [25]. Известно, что фермент RdRp высоко консервативен у вирусов с плюс-цепью РНК-генома, у которых матрицей для синтеза белка служит непосредственно мРНК. Фармакологическое ингибирование активности RdRp возможно с помощью ряда соединений, относящихся к классу аналогов нуклеозидов. Они широко используются для терапии ВИЧ-инфекции и других возбудителей вирусных инфекционных заболеваний, предотвращая репликацию вируса в зараженных клетках. К классу таких лекарственных средств относятся ремдесивир и молнупиравир [26]. Оба препарата подавляют репликацию вируса, но механизм их действия различен. Так, ремдесивир выступает в качестве аналога аденозинтрифосфата и встраивается в формирующиеся РНК-цепи с помощью фермента РНК-зависимой РНК-полимеразы вируса. В результате происходит обрыв цепи во время репликации вирусной РНК [27]. Напротив, молнупиравир фосфорилируется в клетке с образованием фармакологически активного рибонуклеозид-трифосфата, который при встраивании в вирусную РНК приводит к накоплению ошибок в геноме вируса. Количество таких «неправильных» оснований увеличивается в течение нескольких циклов, приводя к подавлению репликации и гибели вируса6.
Ингибиторы протеаз
В процессе репликации SARS-CoV-2 синтезируются длинные полипептиды, которые в ходе процессинга разрезаются на составляющие вирусные белки с помощью двух протеаз: основной протеазы (3CLpro) и PL-протеазы (PLpro) (рис. 2).
Управлением по контролю за качеством продуктов питания и лекарственных средств США (Food and Drug Administration, FDA) был разрешен нирматрелвир в комбинации с ритонавиром для экстренного применения у лиц с высоким риском заражения SARS-CoV-2 под торговым названием «Паксловид» (Paxlovid, Pfizer) [11]. Нирматрелвир является ингибитором основной протеазы вируса (3CLpro), блокирующим активность фермента и останавливающим репликацию вируса. Паксловид также содержит низкие дозы ритонавира — ингибитора протеазы ВИЧ-1 и цитохрома P450 3A, действующего как фармакокинетический усилитель нирматрелвира [28].
Еще одним ингибитором 3CLpro, который недавно получил разрешение на экстренное использование в Японии, является S-217622 (энситрелвир, Xocova), нековалентный непептидный ингибитор, разработанный с помощью подходов вычислительной и медицинской химии. Энситрелвир и другие молекулы, находящиеся на различных стадиях разработки, вскоре могут стать альтернативой паксловиду и откроют возможности для комбинаторной терапии [28–30].
В настоящее время продолжается поиск ингибиторов белка nsp3, который содержит домен папаин-подобной протеазы (PLpro). Показано, что ингибирование PLpro значительно снижает вирусную нагрузку в модельном эксперименте с вирусом SARS-CoV-2. В настоящее время определены кристаллические структуры комплексов с тридцатью различными низкомолекулярными ингибиторами, показавшими свою эффективность в эксперименте. Это позволяет предположить, что PLpro является оптимальной мишенью для противовирусных препаратов следующего поколения [16].
Резистентность вируса SARS-CoV-2
Вирусы, подобные SARS-CoV-2, склонны к наследственной эволюции при адаптации к новым хозяевам под воздействием одной или нескольких мутаций. Это привело к появлению множества новых вариантов с характеристиками, отличными от предковых штаммов. Новые варианты эффективно распространялись в популяции, а применяемые ранее препараты, в первую очередь моноклональные антитела и иммуноглобулины, стали неэффективны7.
Значимость проблемы и риск отбора таких вариантов значительно повышаются у пациентов с ослабленным иммунитетом, которые склонны к длительному инфицированию ввиду сниженной способности организма подавлять репликацию вируса. Появились первые данные о выявлении мутаций резистентности SARS-CoV-2 у иммунокомпрометированных пациентов к ремдесивиру [31] и новому классу препаратов — паксловиду8 [32][33].
Адаптивные мутации в геноме вируса могут изменить его патогенный потенциал. Даже изменение одной аминокислоты может существенно нарушить способность вируса уклоняться от иммунной системы и усложнить разработку лекарственных средств против него. В последнее время значительно увеличилось количество исследований, посвященных изучению резистентности SARS-CoV-2 с применением генотипических и/или фенотипических методов, результаты которых указывают на актуальность данной проблемы [34][35]. Результаты генотипирования позволяют установить мутационный профиль вируса [36]. Фенотипический метод исследования дает возможность на культуре клеток Vero рассчитать концентрацию лекарственного средства, необходимую для подавления репликации SARS-CoV-2 на 50% (показатель IC50), а также сравнить эффективность действия лекарственного средства на модели вируса дикого типа и при наличии мутаций в его геноме [37]. Мутации в зависимости от кратности изменения эффективности концентрации применяемого лекарственного средства, необходимого для подавления репликации вируса с резистентностью, подразделяются на несколько групп по уровню снижения восприимчивости вируса: сильные — более чем в 100 раз; умеренные — в 25–99 раз; средние — в 5–25 раз; слабые — в 5 раз и менее9.
В работе P.L. Tzou с соавт. [36] было предложено классифицировать мутационные изменения в участках 3CLpro и RdRp, как потенциальные мутации лекарственной устойчивости в случае, если они соответствовали одному из следующих трех критериев: связаны с 2,5-кратным или более высоким снижением чувствительности в биохимическом анализе или в культуре клеток; отобраны в ходе эксперимента при исследовании in vitro; обнаружены у пациентов, в схемы лечения которых включены ингибиторы 3CLpro или RdRp.
Мутации в геноме SARS-CoV-2, ассоциированные с резистентностью к ингибиторам РНК-зависимой РНК-полимеразы
В работе A. Szemiel с соавт. [37] в экспериментах in vitro с SARS-CoV-2 показано, что специфические мутационные изменения в nsp12, основной субъединице RdRp, влияют на эффективность ремдесивира. Значение показателя EC50 (полумаксимальная эффективная концентрация) увеличилось с 0,01 до 0,06 мкМ в случае наличия аминокислотных замен P480L или V557L в nsp12. В случае отсутствия ремдесивира продемонстрирована менее эффективная репликация этих изолятов и существенно более низкая жизнеспособность [37]. Еще 12 изолятов, культивируемых в клетках Vero в присутствии ремдесивира в концентрации 1 или 2,5 мкМ, обладали выраженным цитопатическим действием, что свидетельствует об активной репликации вируса в клетке. Кроме того, еще два изолята реплицировались в присутствии ремдесивира в концентрации 7,5 мкМ. Выявлено четырехкратное увеличение показателя IC50 по сравнению с исходным штаммом. В условиях культивирования тех же вирусов без ремдесивира показатель IC50 оставался сопоставимым с родительским штаммом. Эти эксперименты in vitro указывают на появление специфических мутаций устойчивости к ремдесивиру в присутствии препарата.
В другом исследовании продемонстрировано снижение эффективности ремдесивира при замене глутамина на аспартат в позиции 802 (E802D, nsp12). Замены появлялись только в изолятах, пассируемых в присутствии ремдесивира, и отсутствовали в вирусах дикого типа [38]. Культивирование вируса в присутствии ремдесивира в концентрации 0,01 мкМ на протяжении 10 пассажей привело к появлению мутаций E796G и C799F (nsp12) и снижению эффективности действия препарата. Культивирование рекомбинантных вирусов с аминокислотными заменами в позициях E796G, C799F, E802D, D484Y, F480L и F480L/V557L (nsp12) в присутствии ремдесивира также оказало существенное влияние на эффективность препарата. Однако скорость заражения чувствительных клеток мутантными вирусами, устойчивыми к ремдесивиру, за исключением изолята с заменой D484Y, была значительно выше, чем у вируса дикого типа в присутствии ремдесивира. Такие мутантные варианты более эффективно инфицируют в присутствии ремдесивира новые чувствительные клетки по сравнению с вирусами дикого типа [39].
Опубликованы результаты и клинических наблюдений. Так, точечная мутация D484Y в пальцевом субдомене RdRp вируса была обнаружена у 76-летней женщины с В-клеточным иммунодефицитом и персистирующей виремией SARS-CoV-2 после 5-дневного курса лечения ремдеcивиром [39].
Еще один случай развития резистентности описан у пациента с ослабленным иммунитетом и приобретенным В-клеточным иммунодефицитом, у которого развилось вялотекущее, затяжное течение COVID-19. Терапия ремдесивиром вызвала временный вирусологический ответ, но ее течение осложнилось возобновлением выделения вируса с высокой вирусной нагрузкой. Полногеномное секвенирование выявило мутацию E802D в РНК-зависимой РНК-полимеразе nsp12, которой не было в образцах, полученных до начала лечения или в течение первых 5 сут терапии [31].
Новые мутации, обеспечивающие устойчивость к препаратам против SARS-CoV-2, имеют важные клинические последствия. Описаны первые случаи развития независимо приобретенной мутации в РНК-зависимой РНК-полимеразе de novo V792I в открытой рамке считывания, кодирующей nsp12, у двух реципиентов почечного трансплантата после воздействия ремдесивира [40]. Также было показано, что замена V792I быстро формировалась in vitro в присутствии возрастающих концентраций ремдесивира и приводила к увеличению IC50 ремдесивира в 2,6 раза [41]. Представленные данные подчеркивают необходимость дополнительных усилий по выявлению мутаций резистентности и устранению их клинических последствий в группах компрометированных пациентов.
Мутации в геноме SARS-CoV-2, ассоциированные с резистентностью к ингибиторам основной протеазы
Разработка ингибитора основной протеазы (паксловид) — большое достижение в лечении COVID-19, поскольку его можно принимать перорально. Однако одним из важных остается вопрос лекарственной резистентности SARS-CoV-2 к применяемым или разрабатываемым препаратам.
Появление вариантов SARS-CoV-2 с мутационными изменениями в области 3CLpro вызвало озабоченность по поводу потенциальной лекарственной устойчивости. Так, в работе Y. Hu с соавт. [43] было идентифицировано 100 встречающихся в природе мутаций 3CLpro, расположенных в сайте связывания нирматрелвира, среди которых 20 аминокислотных замен, включая S144M/F/A/G/Y, M165T, E166G, H172Q/F и Q192T/S/L/A/I/P/H/V/W/C/F, оказывают прямое влияние на устойчивость к нирматрелвиру (>10-кратное увеличение), который входит в состав лекарственного средства паксловид.
Исследования in vitro, выполненные компанией Pfizer, показали, что у нирматрелвир-селектированных вирусов SARS-CoV-2 имеется как единичная замена S144A, так и замены в следующих комбинациях: T21I+S144A и T21I+S144A+T304I10. Аминокислотная замена H172Y определена как мутация, вызывающая наибольшее снижение активности нирматрелвира, с 233-кратным увеличением значения константы диссоциации11. Однако, учитывая пониженную ферментативную активность вируса с мутацией H172Y по сравнению с вирусом дикого типа и повышенную ферментативную активность вируса с мутацией Q189E, было предположено, что двойной мутант H172Y+Q189E может восстановить сниженную ферментативную активность лекарственно-устойчивого мутанта H172Y. Показано, что двойная мутация H172Y+Q189E приводила как к повышению ферментативной активности, так и к устойчивости к нирматрелвиру [43].
Несколько мутаций Y54C, G138S, L167F, Q192R и A194S были выявлены в эксперименте in vitro как обеспечивающие резистентность к ингибиторам протеазы — 3CLpro (нирматрелвиру), при этом сочетание аминокислотных замен Y54C и L167F в комбинации с P132H в генной сигнатуре варианта омикрон повышает устойчивость к ингибиторам протеазы, что подчеркивает их потенциальную значимость [42]. Необходимо отметить, что замены в позиции 132 (P132L/S) были выявлены также в вирусных изолятах, полученных от четырех пациентов, получавших лечение препаратом паксловид12.
В исследовании, выполненном Y. Zhou с соавт. [44], установлена сочетанная двойная мутация L50F+E166V, которая приводила к 80-кратному снижению чувствительности к нирматрелвиру. В другом исследовании было показано, что мутация E166V помимо 100-кратного увеличения IC50 нирматрелвира, также обеспечивала перекрестную устойчивость к энситрелвиру [45]. Кроме того, мутации L50F и E166V были выявлены в вирусных изолятах от нескольких лиц у людей, получавших лечение препаратом паксловид13. В совокупности все описанные выше данные указывают на то, что мутация E166V обеспечивает устойчивость к препаратам-ингибиторам основной протеазы.
Степень снижения чувствительности SARS-CoV-2 к противовирусным препаратам различна и зависит от конкретных изменений в молекулярных мишенях препаратов. Например, по имеющимся опубликованным данным, с появлением замены S861G в белке nsp12 снижается чувствительность вируса к ремдесивиру в 25–99 раз (табл. 1). При использовании паксловида (нирматрелвир/ритонавир) спектр таких мутаций значительно шире. Так, мутации, обнаруженные в ORF1ab (nsp5:S144A, nsp5:Q189K, nsp5:H172Y, nsp5:E166A и nsp5:F140A) приводят к умеренному или высокому снижению чувствительности к паксловиду (нирматрелвир/ритонавир).
Таблица 1. Мутации ORF1ab вируса SARS-CoV-2, определяющие резистентность к ремдесивиру и паксловиду, и их распространенность в мире и в странах Европейского союза
Table 1. SARS-CoV-2 ORF1ab mutations conferring resistance to remdesivir and paxlovid and the prevalence of these mutations in the world and in the European Union
Мутации ORF1ab ORF1ab mutations | Противовирусные препараты* Antivirals* | Частота распространенности в мире, % Prevalence in the world, % | Частота распространенности в странах Европейского союза, % Prevalence in the European Union, % | ||
Участки Sites | Позиции Positions | Ремдесивир Remdesivir | Паксловид Paxlovid | ||
nsp5 | G15S | – | 4,4 | 0,2 | 0,13 |
Y54A | – | 23,6 | 0 | 0 | |
T135S | – | 3,5 | 0 | 0 | |
F140A | – | 39 | 0 | 0 | |
S144A | – | 91,9 | 0 | 0 | |
H164N | – | 6,4 | 0,03 | 0 | |
E166A | 10,4 | 33,4 | 0 | 0 | |
H172Y | – | 233 | 0 | 0 | |
Q189K | – | 65,4 | 0 | 0 | |
D248E | – | 3,7 | 0 | 0 | |
nsp12 | V166A | – | – | 0,01 | 0,01 |
N198S | 10,4 | – | 0,01 | 0 | |
F480L | 3,8 | – | 0 | 0 | |
D484Y | 3,1 | – | 0 | 0 | |
V557L | 5,7 | – | 0 | 0 | |
V792I | 8 | – | 0 | 0 | |
E796G | 2,6 | – | 0 | 0 | |
C799F | 11,5 | – | 0 | 0 | |
C799R | 2,7 | – | 0 | 0 | |
E802D | 7,3 | – | 0 | 0 | |
E802A | 3,9 | – | 0 | 0 | |
S861G | 29,7 | – | 0 | 0 | |
S861A | 2,8 | – | 0 | 0 | |
Таблица подготовлена авторами по материалам Европейского центра по профилактике и контролю заболеваний14 / The table is adapted by the authors from the materials of the European Centre for Disease Prevention and Control14
Примечание. * — цвет ячеек столбца указывает на уровень снижения восприимчивости вируса к препаратам: красный цвет — в более чем в 100 раз; оранжевый — в 25–99 раз; розовый — в 5–25 раз, зеленый — в 5 раз и менее. «– » — отсутствие данных.
Note.* Cell colours indicate the extent of reduction in susceptibility to antiviral treatment: red, ≥100-fold reduction; orange, 25–99-fold reduction; yellow, 5–25-fold reduction; green, <5-fold reduction; and dashes (–) indicate the absence of data.
Важно отметить и тот факт, что многие варианты вируса с мутациями, влияющими на эффективность применения лекарственных препаратов, существовали до их применения, что было доказано методом филогенетического анализа [33]. Следовательно, эти варианты вируса попадают в человеческую популяцию и способны к дальнейшему распространению.
В целом на данном этапе представленность мутаций резистентности невелика в популяции вируса SARS-CoV-2. Например, исследование 13446588 полногеномных последовательностей вируса SARS-CoV-2, депонированных в общедоступных базах данных до июня 2022 г., показало, что мутации устойчивости к ингибитору 3CLpro остаются редкими и наблюдаются в основном спорадические случаи на протяжении года после начала применения паксловида. Лишь 0,5% проанализированных последовательностей (n=67095) имели такие мутации [34].
Наиболее уязвимой группой являются пациенты с иммуносупрессией. Например, выявлен случай развития лекарственной резистентности у 65-летнего мужчины со злокачественными новообразованиями в анамнезе. У данного пациента, инфицированного SARS-CoV-2, на протяжении 190 сут от момента первичного выявления антигена и РНК вируса проводилось количественное определение уровней антигена и вирусной нагрузки в назофарингеальных мазках. Было также выполнено полногеномное секвенирование вируса для изучения механизмов лекарственной устойчивости. На протяжении всего периода наблюдения пациенту проводили терапию несколькими противовирусными препаратами и антителами, включая сотровимаб, ремдесивир, паксловид (нирматрелвир/ритонавир) и молнупиравир. Первоначально уровни вирусного антигена снизились после лечения. Однако после того, как повторно появились маркеры вируса в мазках (антиген и РНК), у пациента не наблюдалось вирусологического ответа. Анализ вирусного генома выявил единственный субвариант омикрона (BA.1.1), который эволюционировал в организме хозяина в ходе прогрессирования заболевания. Вирус приобрел множественные мутации устойчивости к нирматрелвиру (3CLpro — E166 A/V), сотровимабу (S-белок — P337L и E340K) и ремдесивиру (RdRp — V166L). Благодаря мутациям SARS-CoV-2 получил преимущество в репликации по сравнению с дикими штаммами в присутствии лекарственных препаратов, и такой вид лекарственной устойчивости можно отнести к приобретенной [33].
Таким образом, в отношении COVID-19 современные противовирусные препараты показали клиническую эффективность. В ближайшее время, вероятно, не стоит ожидать значительного увеличения уровня резистентности вируса и повышения необходимости выполнения анализа на генотипическую резистентность до начала лечения пациентов. Но существует и ряд причин, по которым следует продолжать мониторинг резистентности к ингибиторам 3CLpro и RdRp. Во-первых, наиболее частая мутация G15S (встречаемость — 0,2% в мире и 0,13% в странах Европейского союза) доминирует в отдельных генетических вариантах. Это позволяет предположить, что эти мутации не влияют на приспособляемость вируса и могут эффективно передаваться. Следует иметь в виду, что G15S присутствовала в последовательностях вируса до начала клинического использования паксловида, что свидетельствует о спонтанном возникновении мутации, а не отборе при использовании данного препарата. Несмотря на высокое селективное давление, эта мутация может распространяться в популяции [46]. Во-вторых, низкая распространенность мутаций связана и с достаточно ограниченным использованием паксловида и ремдесивира. Нельзя исключить вероятность того, что с увеличением использования противовирусных препаратов в будущем может произойти отбор лекарственно-устойчивых штаммов, особенно если препараты будут применяться в виде монотерапии [47]. В-третьих, резистентность к противовирусным препаратам может возникнуть внезапно. Например, в 2008 г. при лечении сезонного гриппа А (H1N1) произошел значительный всплеск устойчивости к осельтамивиру: с <10% в сезоне 2006–2007 гг. до почти 100% в сезоне 2008–2009 гг., хотя препарат был впервые представлен в конце 1990-х гг. [48]. В-четвертых, важным фактором являются пациенты с иммуносупрессией, у которых SARS-CoV-2 может сохраняться в течение многих месяцев, и противовирусный препарат может потерять свою эффективность по мере отбора или появления спонтанных мутаций, создавая риск распространения лекарственно-устойчивых штаммов и/или неблагоприятного исхода для самого пациента [49].
ЗАКЛЮЧЕНИЕ
Результаты проведенного анализа свидетельствуют о рисках развития устойчивых форм SARS-CoV-2 к применяемым в настоящее время противовирусным препаратам, среди которых ингибиторы RdRp (ремдесивир и молнупиравир) и протеазы 3CLpro (паксловид). Несмотря на то что частота встречаемости мутаций лекарственной устойчивости к ремдесивиру, молнупиравиру и паксловиду в геноме вируса в мире и в европейском регионе остается на достаточно низком уровне, необходимо принимать во внимание факт небольшого опыта их применения. Поэтому по мере увеличения охвата лечением пациентов с COVID-19 с учетом фактора высокой генетической изменчивости вируса SARS-CoV-2 может произойти рост его резистентных вариантов. Вполне возможно, это потребует разработки принципиально новых противовирусных лекарственных средств и/или использования комбинации препаратов в случае выявления мультирезистентных вариантов SARS-СoV-2.
Так как основные публикации о случаях резистентности в клинической практике касаются преимущественно групп пациентов с иммуносупрессией, на современном этапе в надзоре за резистентностью возбудителя COVID-19 особое внимание должно быть уделено лицам с ослабленным иммунитетом.
В целом представленные данные открывают новые перспективы в наблюдении за эволюцией SARS-CoV-2 и свидетельствуют о необходимости проведения геномного эпидемиологического надзора с анализом потенциальных мишеней действия лекарственных препаратов против SARS-CoV-2 и данных клинического наблюдения пациентов, что может иметь важное значение для выбора тактики лечения COVID-19.
Вклад авторов. Все авторы подтверждают соответствие своего авторства критериям ICMJE. Наибольший вклад распределен следующим образом: Е.Л. Гасич — разработка дизайна статьи, написание, редактирование и критическое обсуждение текста рукописи; Е.Н. Кроткова — разработка дизайна статьи и критическое обсуждение текста рукописи, А.Д. Коско — анализ данных литературы, написание текста рукописи; К.Ю. Булда — анализ данных литературы, оформление рукописи, работа с иллюстративным материалом, А.Г. Красько — разработка дизайна статьи, критическое обсуждение текста рукописи; Е.А. Дорофеева — анализ данных литературы; И.А. Карпов — редактирование и переработка текста рукописи.
Authors’ contributions. All the authors confirm that they meet the ICMJE criteria for authorship. The most significant contributions were as follows. E.L. Gasich designed the study; drafted, edited, and critically discussed the manuscript. E.N. Krotkova designed the study and critically discussed the manuscript. A.D. Kasko analysed literature data and drafted the manuscript. K.Yu. Bulda analysed literature data, drafted the manuscript, and worked with the illustrative material. A.G. Krasko designed the study and critically discussed the manuscript. E.A. Dorofeeva analysed literature data. I.A. Karpov edited and revised the manuscript.
1. https://data.who.int/dashboards/covid19/deaths?n=c
2. COVID-19 Public Health Emergency of International Concern (PHEIC) Global research and innovation forum.
https://www.who.int/publications/m/item/covid-19-public-health-emergency-of-international-concern-(pheic)-global-research-and-innovation-forum
3. WHO Director-General’s opening remarks at the media briefing on COVID-19 — 11 March 2020. https://www.who.int/director-general/speeches/detail/who-director-general-s-opening-remarks-at-the-media-briefing-on-covid-19---11-march-2020
4. https://www.who.int/activities/tracking-SARS-CoV-2-variants
5. https://www.bio.org/policy/human-health/vaccines-biodefense/coronavirus/pipeline-tracker
6. https://www.ema.europa.eu/en/documents/smop-initial/questions-and-answers-refusal-marketing-authorisation-lagevrio-molnupiravir_en.pdf
7. SARS-CoV-2 variant mutations conferring reduced susceptibility to antiviral drugs and monoclonal antibodies: a non-systematic literature review for surveillance purposes. ECDC; 2023. https://op.europa.eu/en/publication-detail/-/publication/30781419-2a9a-11ee-839d-01aa75ed71a1/language-en
8. Backman I. New class of drugs could prevent resistant COVID-19 variants. https://medicine.yale.edu/news-article/new-class-of-drugs-could-prevent-resistant-covid-19-variants/
9. SARS-CoV-2 variant mutations conferring reduced susceptibility to antiviral drugs and monoclonal antibodies: a non-systematic literature review for surveillance purposes. ECDC; 2023. https://op.europa.eu/en/publication-detail/-/publication/30781419-2a9a-11ee-839d-01aa75ed71a1/language-en
10. Fact sheet for healthcare providers: emergency use authorization for PAXLOVIDTM. https://www.fda.gov/media/155050/download
11. Там же.
12. Там же.
13. Там же.
14. SARS-CoV-2 variant mutations conferring reduced susceptibility to antiviral drugs and monoclonal antibodies: a non-systematic literature review for surveillance purposes. ECDC; 2023. https://op.europa.eu/en/publication-detail/-/publication/30781419-2a9a-11ee-839d-01aa75ed71a1/language-en
Список литературы
1. Wu F, Zhao S, Yu B, Chen YM, Wang W, Song ZG, et al. A new coronavirus associated with human respiratory disease in China. Nature. 2020;579(7798):265–9. https://doi.org/10.1038/s41586-020-2008-3
2. Li Y, Tenchov R, Smoot J, Liu C, Watkins S, Zhou Q. A comprehensive review of the global efforts on COVID-19 vaccine development. ACS Cent Sci. 2021;7(4):512–33. https://doi.org/10.1021/acscentsci.1c00120
3. Rahmani K, Shavaleh R, Forouhi M, Disfani HF, Kamandi M, Oskooi RK, et al. The effectiveness of COVID-19 vaccines in reducing the incidence, hospitalization, and mortality from COVID-19: a systematic review and meta-analysis. Front Public Health. 2022;10:873596. https://doi.org/10.3389/fpubh.2022.873596
4. Chaudhuri S, Symons JA, Deval J. Innovation and trends in the development and approval of antiviral medicines: 1987–2017 and beyond. Antiviral Res. 2018;155:76–88. https://doi.org/10.1016/j.antiviral.2018.05.005
5. Padmanabha Das KM. Antiviral drug discovery against pathogens of pandemic concern: advancements in target site identification and structure-based drug development. Front Mol Biosci.2023;10:1165208. https://doi.org/10.3389/fmolb.2023.1165208
6. Alkhamis MA, Moore BR, Perez AM. Phylodynamics of H5N1 highly pathogenic avian influenza in Europe, 2005-2010: potential for molecular surveillance of new outbreaks. Viruses. 2015;7(6):3310–28. https://doi.org/10.3390/v7062773
7. Ghany MG, Doo EC. Antiviral resistance and hepatitis B therapy. Hepatology. 2009;49(5 Suppl):S174–84. https://doi.org/10.1002/hep.22900
8. Wensing AM, Calvez V, Ceccherini-Silberstein F, Charpentier C, Günthard HF, Paredes R, et al. 2022 update of the drug resistance mutations in HIV-1. Top Antivir Med. 2022;30(4):559–74. PMID: 36375130
9. Mushtaq S, Hashmi AH, Khan A, Asad Raza Kazmi SM, Manzoor S. Emergence and persistence of resistance-associated substitutions in HCV GT3 patients failing direct-acting antivirals. Front Pharmacol. 2022;13:894460. https://doi.org/10.3389/fphar.2022.894460
10. Nande A, Hill AL. The risk of drug resistance during long-acting antimicrobial therapy. Proc Biol Sci. 2022;289(1986): 20221444. https://doi.org/10.1098/rspb.2022.1444
11. Malin JJ, Suárez I, Priesner V, Fätkenheuer G, Rybniker J. Remdesivir against COVID-19 and other viral diseases. Clin Microbiol Rev. 2020;34(1):e00162-20. https://doi.org/10.1128/CMR.00162-20
12. Hashemian SMR, Sheida A, Taghizadieh M, Memar MY, Hamblin MR, Bannazadeh Baghi H, et al. Paxlovid (nirmatrelvir/ritonavir): a new approach to Covid-19 therapy? Biomed Pharmacother.2023;162:114367. https://doi.org/10.1016/j.biopha.2023.114367
13. Wang MY, Zhao R, Gao LJ, Gao XF, Wang DP, Cao JM. SARS-CoV-2: structure, biology, and structure-based therapeutics development. Front Cell Infect Microbiol. 2020;10:587269. https://doi.org/10.3389/fcimb.2020.587269
14. Naqvi AAT, Fatima K, Mohammad T, Fatima U, Singh IK, Singh A, et al. Insights into SARS-CoV-2 genome, structure, evolution, pathogenesis and therapies: Structural genomics approach. Biochim Biophys Acta Mol Basis Dis. 2020;1866(10):165878. https://doi.org/10.1016/j.bbadis.2020.165878
15. Safiabadi Tali SH, LeBlanc JJ, Sadiq Z, Oyewunmi OD, Camargo C, Nikpour B, et al. Tools and techniques for Severe Acute Respiratory Syndrome Coronavirus 2 (SARS-CoV-2)/COVID-19 detection. Clin Microbiol Rev. 2021;34(3):e00228-20. https://doi.org/10.1128/CMR.00228-20
16. Calleja DJ, Lessene G, Komander D. Inhibitors of SARS-CoV-2 PLpro. Front Chem. 2022;10:876212. https://doi.org/10.3389/fchem.2022.876212
17. Herlocher ML, Elias S, Truscon R, Harrison S, Mindell D, Simon C, Monto AS. Ferrets as a transmission model for influenza: sequence changes in HA1 of type A (H3N2) virus. J Infect Dis. 2001;184(5):542–6. https://doi.org/10.1086/322801
18. Mansky LM. Forward mutation rate of human immunodeficiency virus type 1 in a T-lymphoid cell line. AIDS Res Hum Retroviruses. 1996;12(4):307–14. https://doi.org/10.1089/aid.1996.12.307
19. Van Egeren D, Novokhodko A, Stoddard M, Tran U, Zetter B, Rogers M, et al. Risk of rapid evolutionary escape from biomedical interventions targeting SARS-CoV-2 spike protein. PLoS One. 2021;16(4):e0250780. https://doi.org/10.1371/journal.pone.0250780
20. Li X, Wang W, Zhao X, Zai J, Zhao Q, Li Y, Chaillon A. Transmission dynamics and evolutionary history of 2019-nCoV. J Med Virol. 2020;92(5):501–11. https://doi.org/10.1002/jmv.25701
21. Motayo BO, Oluwasemowo OO, Olusola BA, Akinduti PA, Arege OT, Obafemi YD, et al. Evolution and genetic diversity of SARS-CoV-2 in Africa using whole genome sequences. Int J Infect Dis. 2021;103:282–7. https://doi.org/10.1016/j.ijid.2020.11.190
22. Rahmah L, Abarikwu SO, Arero AG, Essouma M, Jibril AT, Fal A, et al. Oral antiviral treatments for COVID-19: opportunities and challenges. Pharmacol Rep. 2022;74(6):1255–78. https://doi.org/10.1007/s43440-022-00388-7
23. Duan X, Lacko LA, Chen S. Druggable targets and therapeutic development for COVID-19. Front Chem. 2022;10:963701. https://doi.org/10.3389/fchem.2022.963701
24. Su H, Xu Y, Jiang H. Drug discovery and development targeting the life cycle of SARS-CoV-2. Fundamental Res. 2021;1(2):151–65. https://doi.org/10.1016/j.fmre.2021.01.013
25. Kadam SB, Sukhramani GS, Bishnoi P, Pable AA, Barvkar VT. SARS-CoV-2, the pandemic coronavirus: molecular and structural insights. J Basic Microbiol. 2021;61(3):180–202. https://doi.org/10.1002/jobm.202000537
26. Vangeel L, Chiu W, De Jonghe S, Maes P, Slechten B, Raymenants J, et al. Remdesivir, Molnupiravir and Nirmatrelvir remain active against SARS-CoV-2 Omicron and other variants of concern. Antiviral Res. 2022;198:105252. https://doi.org/10.1016/j.antiviral.2022.105252
27. Gordon CJ, Tchesnokov EP, Feng JY, Porter DP, Götte M. The antiviral compound remdesivir potently inhibits RNA-dependent RNA polymerase from Middle East respiratory syndrome coronavirus. J Biol Chem. 2020;295(15):4773–9. https://doi.org/10.1074/jbc.AC120.013056
28. Shimizu R, Sonoyama T, Fukuhara T, Kuwata A, Matsuzaki T, Matsuo Y, Kubota R. Evaluation of the drug–drug interaction potential of ensitrelvir fumaric acid with cytochrome P450 3A substrates in healthy Japanese adults. Clin Drug Investig. 2023;43(5):335–46. https://doi.org/10.1007/s40261-023-01265-8
29. Unoh Y, Uehara S, Nakahara K, Nobori H, Yamatsu Y, Yamamoto S, et al. Discovery of S-217622, a noncovalent oral SARS-CoV-2 3CL protease inhibitor clinical candidate for treating COVID-19. J Med Chem. 2022;65(9):6499–512. https://doi.org/10.1021/acs.jmedchem.2c00117
30. Bege M, Borbás A. The design, synthesis and mechanism of action of paxlovid, a protease inhibitor drug combination for the treatment of COVID-19. Pharmaceutics. 2024;16(2):217. https://doi.org/10.3390/pharmaceutics16020217
31. Gandhi S, Klein J, Robertson A, Peña-Hernández MA, Lin MJ, Roychoudhury P, et al. De novo emergence of a remdesivir resistance mutation during treatment of persistent SARS-CoV-2 infection in an immunocompromised patient: a case report. Nat Commun. 2022;13(1):1547. https://doi.org/10.1038/s41467-022-29104-y
32. Moghadasi SA, Heilmann E, Khalil AM, Nnabuife C, Kearns FL, Ye C, et al. Transmissible SARS-CoV-2 variants with resistance to clinical protease inhibitors. Sci Adv. 2023;9(13):eade8778. https://doi.org/10.1126/sciadv.ade8778
33. Hirotsu Y, Kobayashi H, Kakizaki Y, Saito A, Tsutsui T, Kawaguchi M, et al. Multidrug-resistant mutations to antiviral and antibody therapy in an immunocompromised patient infected with SARS-CoV-2. Med. 2023;4(11):813–824.e4. https://doi.org/10.1016/j.medj.2023.08.001
34. Ip JD, Wing-Ho Chu A, Chan WM, Cheuk-Ying Leung R, Umer Abdullah SM, Sun Y, Kai-Wang To K. Global prevalence of SARS-CoV-2 3CL protease mutations associated with nirmatrelvir or ensitrelvir resistance. EBioMedicine. 2023;91:104559. https://doi.org/10.1016/j.ebiom.2023.104559
35. Vitiello A. Sars-Cov-2 and risk of antiviral drug resistance. Ir J Med Sci. 2022;191(5):2367–8. https://doi.org/10.1007/s11845-021-02820-y
36. Tzou PL, Tao K, Sahoo MK, Kosakovsky Pond SL, Pinsky BA, Shafer RW. Sierra SARS-CoV-2 sequence and antiviral resistance analysis program. J Clin Virol. 2022;157:105323. https://doi.org/10.1016/j.jcv.2022.105323
37. Szemiel AM, Merits A, Orton RJ, MacLean O, Wickenhagen A, Lieber G, et al. In vitro evolution of remdesivir resistance reveals genome plasticity of SARS-CoV-2. Preprint-Biorxiv. 2021;ID:ppbiorxiv-429199 https://doi.org/10.1101/2021.02.01.429199
38. Checkmahomed L, Carbonneau J, Du Pont V, Riola NC, Perry JK, Li J, et al. In vitro selection of remdesivir-resistant SARS-CoV-2 demonstrates high barrier to resistance. Antimicrob Agents Chemother. 2022;66(7):e0019822. https://doi.org/10.1128/aac.00198-22
39. Martinot M, Jary A, Fafi-Kremer S, Leducq V, Delagreverie H, Garnier M, et al. Emerging RNA-dependent RNA polymerase mutation in a remdesivir-treated B-cell immunodeficient patient with protracted coronavirus disease 2019. Clin Infect Dis. 2021;73(7):e1762–5. https://doi.org/10.1093/cid/ciaa1474
40. Hogan JI, Duerr R, Dimartino D, Marier C, Hochman S, Mehta S, et al. Remdesivir resistance in transplant recipients with persistent COVID-19. Clin Infect Dis. 2023;76(2):342–5. https://doi.org/10.1093/cid/ciac769
41. Stevens LJ, Pruijssers AJ, Lee HW, Gordon CJ, Tchesnokov EP, Gribble J, et al. Mutations in the SARS-CoV-2 RNA-dependent RNA polymerase confer resistance to remdesivir by distinct mechanisms. Sci Transl Med. 2022;14(656):eabo0718. https://doi.org/10.1126/scitranslmed.abo0718
42. Heilmann E, Costacurta F, Moghadasi SA, Ye C, Pavan M, Bassani D et al. SARS-CoV-2 3CLpro mutations selected in a VSV-based system confer resistance to nirmatrelvir, ensitrelvir, and GC376. Sci Transl Med. 2023;15(678):eabq7360. https://doi.org/10.1126/scitranslmed.abq7360
43. Hu Y, Lewandowski EM, Tan H, Zhang X, Morgan RT, Zhang X, et al. Naturally occurring mutations of SARS-CoV-2 main protease confer drug resistance to nirmatrelvir. ACS Cent Sci. 2023;9(8):1658–69.https://doi.org/10.1021/acscentsci.3c00538
44. Zhou Y, Gammeltoft KA, Ryberg LA, Pham LV, Tjørnelund HD, Binderup A, et al. Nirmatrelvir-resistant SARS-CoV-2 variants with high fitness in an infectious cell culture system. Sci Adv. 2022;8(51):eadd7197. https://doi.org/10.1126/sciadv.add7197
45. Iketani S, Mohri H, Culbertson B, Hong SJ, Duan Y, Luck MI, et al. Multiple pathways for SARS-CoV-2 resistance to nirmatrelvir. Nature. 2023;613(7944):558–64. https://doi.org/10.1038/s41586-022-05514-2
46. Sjaarda CP, Lau L, Simpson JT, Fattouh R, Biondi MJ, Maguire F, et al. Prevalence of low-frequency, antiviral resistance variants in SARS-CoV-2 isolates in Ontario, Canada, 2020–2023. JAMA Netw Open. 2023;6(7):e2324963. https://doi.org/10.1001/jamanetworkopen.2023.24963
47. Kozlov M. COVID drug Paxlovid was hailed as a game-changer. What happened? Nature. 2023;613(7943):224–5. https://doi.org/10.1038/d41586-022-04576-6
48. Weinstock DM, Zuccotti G. The evolution of influenza resistance and treatment. JAMA. 2009;301(10):1066–9. https://doi.org/10.1001/jama.2009.324
49. Guilbaud R, Franco Yusti AM, Leducq V, Zafilaza K, Bridier-Nahmias A, Todesco E, et al. Higher levels of SARS-CoV-2 genetic variation in immunocompromised patients: a retrospective case-control study. J Infect Dis. 2023:jiad499. https://doi.org/10.1093/infdis/jiad499
Об авторах
Е. Л. ГасичБеларусь
Гасич Елена Леонидовна, д-р биол. наук, доц.
ул. Казинца, д. 50, г. Минск, 220099
Е. Н. Кроткова
Беларусь
Кроткова Елена Николаевна, канд. мед. наук, доц.
ул. Долгиновский тракт, д. 157, г. Минск, 220080
А. Д. Коско
Беларусь
Коско Анастасия Дмитриевна
ул. Казинца, д. 50, г. Минск, 220099
К. Ю. Булда
Беларусь
Булда Кирилл Юрьевич
ул. Казинца, д. 50, г. Минск, 220099,
А. Г. Красько
Беларусь
Красько Анатолий Геннадиевич, канд. мед. наук
ул. Казинца, д. 50, г. Минск, 220099
Е. А. Дорофеева
Беларусь
Дорофеева Елизавета Алексеевна
ул. Голубева, д. 25, г. Минск, 220117
И. А. Карпов
Беларусь
Карпов Игорь Александрович, д-р мед. наук, проф., член-корр. Национальной академии наук Республики Беларусь
пр. Дзержинского, д. 83, г. Минск, 220083
Дополнительные файлы
Рецензия
Для цитирования:
Гасич Е.Л., Кроткова Е.Н., Коско А.Д., Булда К.Ю., Красько А.Г., Дорофеева Е.А., Карпов И.А. Проблема лекарственной устойчивости вируса SARS-CoV-2 к препаратам прямого противовирусного действия. БИОпрепараты. Профилактика, диагностика, лечение. 2024;24(4):415-427. https://doi.org/10.30895/2221-996X-2024-569
For citation:
Gasich E.L., Krotkova E.N., Kasko A.D., Bulda K.Yu., Krasko A.G., Dorofeeva E.A., Karpov I.A. The problem of SARS-CoV-2 virus resistance to direct-acting antivirals. Biological Products. Prevention, Diagnosis, Treatment. 2024;24(4):415-427. (In Russ.) https://doi.org/10.30895/2221-996X-2024-569


































